Het menselijk genoom bevat tienduizenden genen: minuscule DNA-strengen die coderen voor de eigenschappen en kenmerken die ieder van ons uniek maken. Elk gen krijgt een naam en een alfanumerieke code — ook wel een symbool genoemd — die wetenschappers gebruiken om onderzoek te coördineren. Maar het afgelopen jaar werden de symbolen van 27 genen hernoemd omdat Microsoft Excel ze foutief interpreteert als datums.
Dat is niet zo'n futiliteit als op het eerste gezicht lijkt. Excel is immers een mastodont in de spreadsheetwereld en wordt door wetenschappers vaak gebruikt om hun werk te beheren en zelfs klinische onderzoeken uit te voeren. De standaardinstellingen van Excel zijn echter ontworpen met meer alledaagse toepassingen in gedachten. Wanneer een gebruiker een alfanumeriek gensymbool zoals MARCH1 in een spreadsheet invoert — de afkorting van Membrane Associated Ring-CH-Type Finger 11 — dan zet Excel dit automatisch om in een datum: 1-Mar.
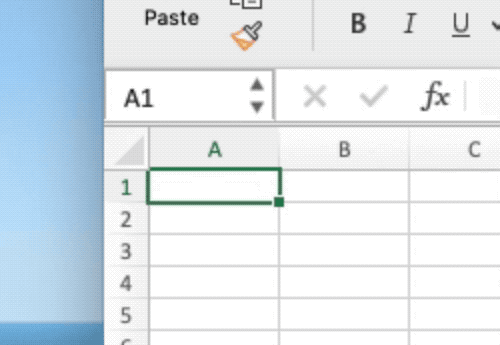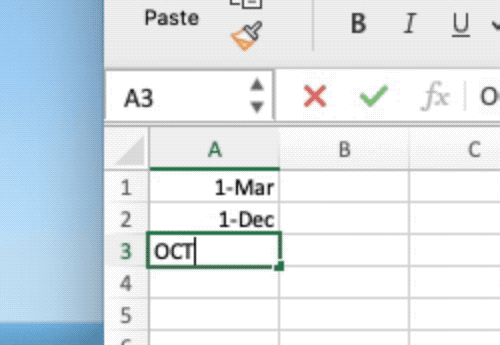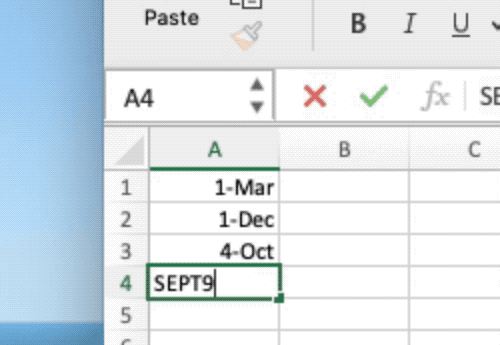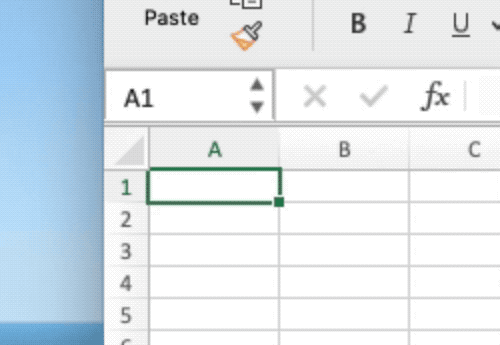
Dit zijn buitengewoon frustrerende — zelfs gevaarlijke — corrupte gegevens die wetenschappers met de hand moeten doorzoeken om ze te herstellen. Het is ook verrassend wijdverbreid en sluipt zelfs in wetenschappelijk werk dat door experten werd nagekeken. Een studie uit 2016 onderzocht genetische gegevens die werden gedeeld in 3.597 wetenschappelijke publicaties en ontdekte dat ongeveer een vijfde was getroffen door Excel-fouten.
Het probleem kan ook niet makkelijk opgelost worden. Excel biedt geen optie om de automatische aanpassingen uit te schakelen. De enige manier om dit te vermijden, is door het gegevenstype voor afzonderlijke kolommen te wijzigen2. Maar zelfs dan nog kan een wetenschapper zijn gegevens corrigeren en daarna exporteren als een CSV-bestand waarin de opmaak niet wordt meegenomen. Of een andere wetenschapper laadt de gegevens in zonder de juiste opmaak, waardoor gensymbolen weer in datums veranderen. Het eindresultaat is dat — hoewel ervaren Excel-gebruikers dit probleem kunnen vermijden — er gemakkelijk fouten kunnen geïntroduceerd worden.
Er kwam echter hulp uit de hoek van de wetenschappelijke instantie die verantwoordelijk is voor het standaardiseren van de namen van genen: de HUGO Gene Nomenclature Committee (HGNC). Deze publiceerde in 2020 nieuwe richtlijnen3 voor de naamgeving van genen, inclusief voor "symbolen die van invloed zijn op het verwerken en ophalen van gegevens". Daarin zeggen ze dat menselijke genen van nu af aan een naam zullen krijgen die niet gevoelig is voor de automatische opmaak van Excel. Daardoor is bijvoorbeeld het symbool MARCH1 nu MARCHF1 geworden, en werd SEPT1 hernoemd naar SEPTIN1.
Opgave
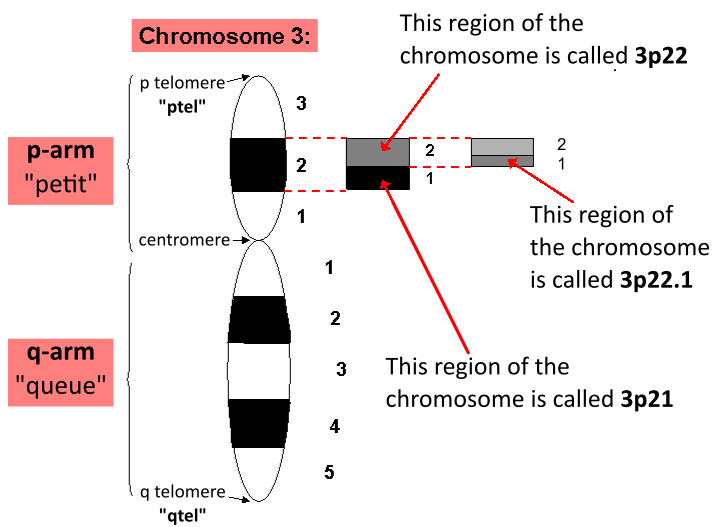
Het menselijk genoom is de erfelijke informatie die als DNA4 gecodeerd zit in elke cel van het menselijk lichaam. In de celkern zit het DNA gegroepeerd in 23 paar chromosomen. Door een microscoop bekeken heeft zo'n chromosoom een karakteristieke vierarmige structuur: een paar zusterchromatiden die aan elkaar zijn bevestigd op het centromeer. De korte armen worden de p-armen genoemd (van het Franse woord petit; wat klein betekent) en de langere armen worden de q-armen genoemd (de letter q volgt gewoon op p in het alfabet; als alternatief wordt soms ook gezegd dat q staat voor het Franse woord queue, wat staart betekent).

Een gen is een gebied binnen het DNA dat omgezet wordt naar een eiwit dat uiteindelijk een aantal functies binnen de cel zal uitvoeren. Het menselijk genoom bevat naar schatting 20.000 genen die voor eiwitten coderen. Om de positie van een gen op een chromosoom te beschrijven, wordt elke arm (of telomeer) van een chromosoom verder opgedeeld in regio's die op hun beurt uit banden (en bijhorende subbanden) bestaan. Zo duidt positie 3p22.1 op chromosoom 3, de p-telomeer, regio 2, band 2 en tenslotte subband 1. Naast de 22 genummerde chromosomen bestaan er nog 2 chromosomen die door een letter geïdentificeerd worden: X en Y.
Het tekstbestand genes.csv5 bevat informatie over een aantal genen van het menselijk genoom. De eerste regel van het bestand bevat een hoofding. Elke volgende regel bevat informatie over één gen, bestaande uit de volgende zeven velden die van elkaar gescheiden worden door een puntkomma (;):
-
symbol: Uniek symbool dat gebruikt wordt om het gen te identificeren.
-
name: Volledige naam van het gen.
-
status: Toestand van de record over het gen in de HGNC databank6. Dit kan ofwel Approved of Entry Withdrawn zijn.
-
location: Positie van het gen op een chromosoom.
-
date_approved: Datum waarop de informatie over het gen voor het eerst werd goedgekeurd.
-
date_modified Datum waarop de informatie over het gen voor het laatste werd bijgewerkt.
-
entrez_id Unieke identifier van het gen in de Entrez7 databank.
Gevraagd wordt om — gebruikmakend van de teksteditors vi
of vim — een reeks commando's op te stellen die
achtereenvolgens de volgende opdrachten uitvoeren. Probeer voor elke
opdracht zo weinig mogelijk commando's te gebruiken en zorg er voor dat
elk van deze commando's bestaat uit zo weinig mogelijk tekens. De
commando's mogen geen programmeerbare filters (bv. awk)
gebruiken. Alle opdrachten moeten na elkaar uitgevoerd worden. De
opdrachten mogen de eerste regel niet wijzigen, tenzij dit expliciet
anders vermeld staat. Ter controle kan je gebruikmaken van de meegeleverde
bestanden genes.i.csv (ZIP8),
die telkens de inhoud van het bestand bevatten nadat de $$i$$-de opdracht
werd uitgevoerd.
-
Voeg rechts van de kolom location een nieuwe kolom location_sortable toe. Deze kolom bevat dezelfde gegevens als de kolom location, maar als de positie van een gen begint met één cijfer gevolgd door de letter p of q dan moet dit voorafgegaan worden door een voorloopnul (0). Zo moet
symbol;name;status;location;date_approved;date_modified;entrez_id AGPAT5P1;1-acylglycerol-3-phosphate O-acyltransferase 5 pseudogene 1;Approved;Yp11.2;03.02.2010;19.11.2014;646231 ABHD11-AS2;ABHD11 antisense RNA 2;Entry Withdrawn;7q11.23;28.04.2011;01.08.2019; ACACA;acetyl-CoA carboxylase alpha;Approved;17q12;11.09.1989;03.05.2018;31 PXYLP1;2-phosphoxylose phosphatase 1;Approved;3q23;16.09.2004;17.03.2014;92370
bijvoorbeeld omgezet worden naar (genes.1.csv9)
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id AGPAT5P1;1-acylglycerol-3-phosphate O-acyltransferase 5 pseudogene 1;Approved;Yp11.2;Yp11.2;03.02.2010;19.11.2014;646231 ABHD11-AS2;ABHD11 antisense RNA 2;Entry Withdrawn;7q11.23;07q11.23;28.04.2011;01.08.2019; ACACA;acetyl-CoA carboxylase alpha;Approved;17q12;17q12;11.09.1989;03.05.2018;31 PXYLP1;2-phosphoxylose phosphatase 1;Approved;3q23;03q23;16.09.2004;17.03.2014;92370
-
Zet de datums uit de kolom date_approved om van het Europese datumformaat DD.MM.YYYY naar het internationale YYYY-MM-DD formaat. De datums in de kolom date_modified mogen niet aangepast worden. We krijgen dan (genes.2.csv10)
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id AGPAT5P1;1-acylglycerol-3-phosphate O-acyltransferase 5 pseudogene 1;Approved;Yp11.2;Yp11.2;2010-02-03;19.11.2014;646231 ABHD11-AS2;ABHD11 antisense RNA 2;Entry Withdrawn;7q11.23;07q11.23;2011-04-28;01.08.2019; ACACA;acetyl-CoA carboxylase alpha;Approved;17q12;17q12;1989-09-11;03.05.2018;31 PXYLP1;2-phosphoxylose phosphatase 1;Approved;3q23;03q23;2004-09-16;17.03.2014;92370
-
Verwijder alle genen waarvan de inhoud van het veld status gelijk is aan Entry Withdrawn. We krijgen dan (genes.3.csv11)
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id AGPAT5P1;1-acylglycerol-3-phosphate O-acyltransferase 5 pseudogene 1;Approved;Yp11.2;Yp11.2;2010-02-03;19.11.2014;646231 ACACA;acetyl-CoA carboxylase alpha;Approved;17q12;17q12;1989-09-11;03.05.2018;31 PXYLP1;2-phosphoxylose phosphatase 1;Approved;3q23;03q23;2004-09-16;17.03.2014;92370 DECR1;2,4-dienoyl-CoA reductase 1;Approved;8q21.3;08q21.3;1997-07-22;02.12.2016;1666
-
Zet alle voorkomens van de griekse letters alpha, beta en gamma in de volledige naam (name) van een gen om naar hoofdletters. Hierbij mag geen onderscheid gemaakt worden tussen hoofdletters en kleine letters (aLpHa moet dus ook omgezet worden naar ALPHA). We krijgen dan (genes.4.csv12)
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id AGPAT5P1;1-acylglycerol-3-phosphate O-acyltransferase 5 pseudogene 1;Approved;Yp11.2;Yp11.2;2010-02-03;19.11.2014;646231 ACACA;acetyl-CoA carboxylase ALPHA;Approved;17q12;17q12;1989-09-11;03.05.2018;31 PXYLP1;2-phosphoxylose phosphatase 1;Approved;3q23;03q23;2004-09-16;17.03.2014;92370 DECR1;2,4-dienoyl-CoA reductase 1;Approved;8q21.3;08q21.3;1997-07-22;02.12.2016;1666
-
Sorteer het bestand eerst volgens aflopende date_approved en daarna volgens oplopende entrez_id. We krijgen dan (genes.5.csv13)
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id HFE-AS1;HFE antisense RNA 1;Approved;6p22.2;06p22.2;2020-07-22;22.07.2020;108783645 CD84P1;CD84 pseudogene 1;Approved;Xq28;Xq28;2020-07-17;17.07.2020; SRD5A2P1;SRD5A2 pseudogene 1;Approved;Xq28;Xq28;2020-07-17;17.07.2020; OR7E85BP;olfactory receptor family 7 subfamily E member 85B pseudogene;Approved;4p16.1;04p16.1;2020-07-17;17.07.2020;650293
-
Het bestand werd verwerkt met Excel, waardoor alle symbolen die als datum kunnen geïnterpreteerd worden in een verkeerd formaat werden opgeslagen (bijvoorbeeld MARCH1 werd 1-Mar en SEPT5 werd 5-Sep). Vervang het symbool van genen die foutief werden opgeslagen door hun nieuwe symbool. Zo moet 1-Mar bijvoorbeeld MARCHF1 worden en moet 5-Sep omgezet worden in SEPTIN5. Zo moet
symbol;name;status;location;location_sortable;date_approved;date_modified;entrez_id … 10-Mar;MARCHF10 divergent transcript;Approved;17q23.2;17q23.2;2019-06-14;14.08.2019;101927877 … PPP1R35-AS1;PPP1R35 antisense RNA 1;Approved;7q22.1;07q22.1;2019-07-24;24.07.2019;101927632 … 12-Sep;septin 12;Approved;16p13.3;16p13.3;2006-11-13;05.09.2019;124404 … 6-Mar;membrane associated ring-CH-type finger 6;Approved;5p15.2;05p15.2;2005-01-26;14.08.2019;10299 …
bijvoorbeeld omgezet worden naar (genes.6.csv14)
symbol;name;status;location;location_sortable;date_approved_reserved;date_modified;entrez_id … MARCHF10;MARCHF10 divergent transcript;Approved;17q23.2;17q23.2;2019-06-14;14.08.2019;101927877 … PPP1R35-AS1;PPP1R35 antisense RNA 1;Approved;7q22.1;07q22.1;2019-07-24;24.07.2019;101927632 … SEPTIN12;septin 12;Approved;16p13.3;16p13.3;2006-11-13;05.09.2019;124404 … MARCHF6;membrane associated ring-CH-type finger 6;Approved;5p15.2;05p15.2;2005-01-26;14.08.2019;10299 …