Vergelijken van \(g\) Behandelingen
In deze sectie veralgemenen we de methoden uit de vorige sectie. De methoden kunnen ook gezien worden als niet-parametrische tegenhangers van de \(F\)-test uit een one-way ANOVA.
DMH Voorbeeld
Nieuwe (en bestaande) chemische substaties moeten getest worden op genotoxiciteit. De resultaten van genotoxiciteitstesten vormen de basis voor risic0-analyses en de classificatie en labeling van chemische substanties in de EU (Dangerous Substances Directive 67/548/EEC and Regulation (EC) No. 1272/2008). In dat kader werd een studie met 24 ratten opgezet voor het testen van de genotoxiciteit van 1,2-dimethylhydrazine dihydrochloride (DMH). De ratten werden at random verdeeld over vier groepen die een verschillende dagelijkse DMH dosis kregen toegediend (controle, laag, medium, hoog). Na drie weken werden de dieren afgemaakt en werd genotoxiciteit van DMH in de lever bepaald a.d.h.v. een comet assay waarbij DNA strengbreuken worden gevisualiseerd via gel electroforese. De lengte van de comet staart is een proxy voor het aantal strengbreuken. De onderzoekers wensen na te gaan of er verschillen zijn in de DNA schade tengevolge van de DMH dosis. Boxplots van de data worden weergegeven in Figuur 52.
dna <- read_delim("https://raw.githubusercontent.com/statOmics/sbc21/master/data/dna.txt",delim=" ")
dna <- dna %>%
mutate(dose = as.factor(dna$dose))
dna
## # A tibble: 24 × 3
## id length dose
## <chr> <dbl> <fct>
## 1 Rat1 19.3 0
## 2 Rat2 18.9 0
## 3 Rat3 18.6 0
## 4 Rat4 19.0 0
## 5 Rat5 17.1 0
## 6 Rat6 18.8 0
## 7 Rat7 55.4 1.25
## 8 Rat8 59.2 1.25
## 9 Rat9 59.1 1.25
## 10 Rat10 52.1 1.25
## # … with 14 more rows
dna %>%
ggplot(aes(x=dose,y=length,fill=dose))+
geom_boxplot() +
geom_point(position="jitter")
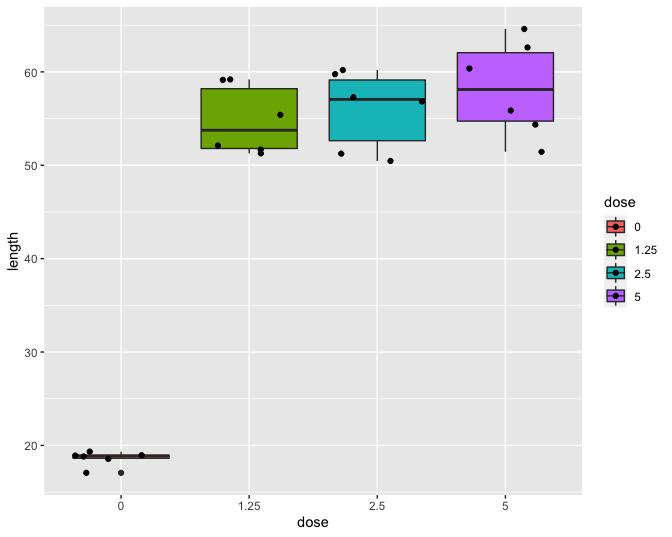
Figuur 52: Boxplot van de comet staart lengte in functie van de DMH dosis.
dna %>%
ggplot(aes(sample=length)) +
geom_qq() +
geom_qq_line() +
facet_wrap(~dose)
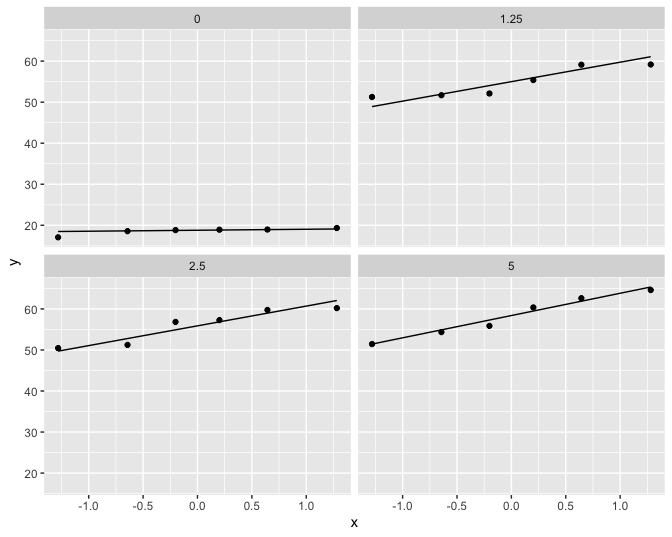
Figuur 53: QQ-plots van de comet staart lengte per DMH dosis.
De boxplots lijken een indicatie te geven dat de controle groep een andere variabiliteit heeft. Merk wel op dat er slechts 6 observaties zijn per groep wat eigenlijk te weinig is om de aannames na te gaan.