Borstkanker dataset
Sotiriou et al. (2006) publiceerden onderzoek naar de moleculaire basis van borstkanker. In de studie hebben de onderzoekers voor een groot aantal borstkanker patiënten klinische variabelen geregistreerd alsook de genexpressie in tumor weefsel gemeten voor duizenden genen m.b.v. microarray technologie. De genexpressie werd gemeten op de tumor biopsie die werd genomen voordat de behandeling werd gestart. De studie is een retrospectieve studie in de zin dat niet werd geëxperimenteerd en dat de genexpressie werd geëvalueerd als gevolg van de blootstelling die de individuen hebben ondergaan in het verleden.
In dit hoofdstuk zullen we een subset van de data gebruiken om de associatie te bestuderen tussen de genexpressie van twee sleutelgenen bij borstkanker: de estrogeen receptor 1 (ESR1) gen, een belangrijke biomerker voor de prognose van de patiënt, en het S100A8 gen dat een prominente rol speelt in de regulatie van inflammatie en immuun respons.
De data is opgeslagen in een tekst bestand met naam brca.csv op de
github repository van de cursus.
brca <- read_csv("https://raw.githubusercontent.com/statOmics/sbc20/master/data/breastcancer.csv")
knitr::kable(head(brca),caption="Overzicht van de variabelen in de borstkanker dataset.",booktabs = TRUE)
| sample\_name | filename | treatment | er | grade | node | size | age | ESR1 | S100A8 |
|---|---|---|---|---|---|---|---|---|---|
| OXFT\_209 | gsm65344.cel.gz | tamoxifen | 1 | 3 | 1 | 2.5 | 66 | 1939.1990 | 207.19682 |
| OXFT\_1769 | gsm65345.cel.gz | tamoxifen | 1 | 1 | 1 | 3.5 | 86 | 2751.9521 | 36.98611 |
| OXFT\_2093 | gsm65347.cel.gz | tamoxifen | 1 | 1 | 1 | 2.2 | 74 | 379.1951 | 2364.18306 |
| OXFT\_1770 | gsm65348.cel.gz | tamoxifen | 1 | 1 | 1 | 1.7 | 69 | 2531.7473 | 23.61504 |
| OXFT\_1342 | gsm65350.cel.gz | tamoxifen | 1 | 3 | 0 | 2.5 | 62 | 141.0508 | 3218.74109 |
| OXFT\_2338 | gsm65352.cel.gz | tamoxifen | 1 | 3 | 1 | 1.4 | 63 | 1495.4213 | 107.56868 |
Data exploratie
In Sectie 4.7 werd de associatie tussen beide genen uitgebreid verkend. Daarin hebben we de genexpressie data eerst log-getransformeerd.
In dit hoofdstuk zullen we om didactische redenen eerst werken met de expressiemetingen op de originele schaal. De expressie van het S100A8 gen wordt weergegeven in Figuur 25. Op de originele schaal zien we drie heel grote outliers. Omwille van didactische redenen worden deze eerst verwijderd uit de dataset. In principe mogen outliers enkel worden verwijderd uit een studie als daar een goede reden voor is. We kunnen op basis van de informatie over de studie echter niet argumenteren waarom de outliers niet representatief zijn, zoals bijvoorbeeld wel het geval zou zijn wanneer zich meetfouten of problemen voordeden m.b.t. deze observaties in de studie. Later in het hoofdstuk zullen we zien hoe we op een correcte wijze alle data kunnen modelleren.
brca %>%
ggplot(aes(x="", y=S100A8)) +
geom_boxplot() +
xlab("") +
ylab("S100A8 expressie")

Figuur 25: Expressie van het S100A8 gen.
Om meerdere variabelen in de borstkanker dataset te bestuderen, kunnen we gebruik maken van de grafische scatterplot matrix voorstelling (zie Figuur 26). Hierbij wordt een matrix met paarsgewijze dotplots voor alle variabelen geproduceerd.
library(GGally)
brcaSubset <- brca %>%
filter(S100A8<2000)
#progress = FALSE zo dat ggpairs niet de vooruitgang print van het plotten
brcaSubset[,-(1:4)] %>% ggpairs(progress = FALSE)

Figuur 26: Scatterplot matrix voor de observaties in de borstkanker dataset na verwijdering van outliers in de S100A8 expressie (merk op dat we deze outliers in principe niet mochten verwijderen uit de dataset).
In de scatterplot matrix zien we bijvoorbeeld dat er een positieve associatie lijkt te zijn tussen de leeftijd (age) en de lymfeknoop status (node; geeft aan of de lymfeknopen al dan niet aangetast zijn en chirurgisch werden verwijderd, node 0: niet aangetast, 1: aangetast). Daarnaast observeren we ook een indicatie voor een negatieve associatie (dalende trend) tussen de ESR1 en S100A8 gen expressie.
In dit hoofdstuk zullen we ons in het bijzonder focussen op de relatie tussen de ESR1 en de S100A8 gen expressie. Een individuele scatterplot met smoother (zie Figuur 27) geeft de associatie tussen beide genen nog beter weer. Smoothers kunnen trends visualiseren tussen variabelen zonder vooraf veronderstellingen te doen over de vorm van het verband en zijn daarom heel erg nuttig bij data exploratie. We zien dat de genexpressie van S100A8 gemiddeld gezien daalt voor patiënten met een hogere expressie van ESR1.
- pipe dataset naar ggplot
- selecteer data
ggplot(aes(x=ESR1,y=S100A8)) - voeg punten toe met
geom_point() - voeg een “smooth line” toe
geom_smooth()
brcaSubset %>%
ggplot(aes(x=ESR1, y=S100A8)) +
geom_point() +
geom_smooth()
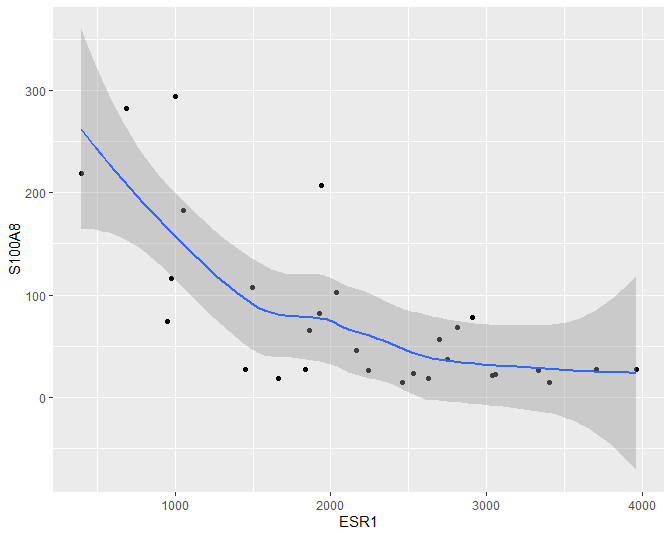
Figuur 27: Scatterplot voor S100A8 expressie in functie van de ESR1 expressie met smoother die het verband tussen beide genen samenvat (na verwijdering van outliers in de S100A8 expressie, merk op dat we deze outliers in principe niet mochten verwijderen uit de dataset).
Model
Op basis van Figuur 27 zien we dat er een relatie is tussen de S100A8 (Y) en ESR1 (X) expressie. De expressiemetingen voor het S100A8 gen zijn echter onderhevig aan ruis onder andere door biologische variabiliteit en technische variabiliteit. Voor een gegeven waarde \(X=x\) neemt de genexpressie \(Y\) dus niet steeds dezelfde waarde aan. Generiek kunnen we de S100A8 gen expressie dus beschrijven als
\[\text{observatie = signaal + ruis.}\]Wiskundig kunnen we dat modelleren als
\[Y_i=g(X_i)+\epsilon_i\]waarbij we de toevallige veranderlijke S100A8 genexpressie voor subject \(i\) (\(Y_i\)) modelleren in functie van de genexpressie van het ESR1 gen (\(X_i\)). Uiteraard is dit verband niet perfect. Dat wordt aangegeven door de foutterm \(\epsilon_i\) die uitdrukt dat observaties \(Y_i\) variëren rond dit verband, m.a.w. het verband modelleert een conditioneel gemiddelde:
\[E[Y_i|X_i=x]=g(x),\]het is de verwachte uitkomst (\(E[Y]\)) bij subjecten met een expressieniveau \(X_i=x\) voor het ESR1 gen.
Zo geeft \(E(Y|X=2400)\) de gemiddelde genexpressie aan van het S100A8 gen voor subjecten die een expressie hebben van 2400 voor het ESR1 gen. Men zou dit gemiddelde bekomen door van alle patiënten in de studiepopulatie, die een ESR1 expressie hebben van 2400, de S100A8 expressie te meten en hier vervolgens het gemiddelde van te nemen. Het gemiddelde \(E(Y|X=x)\) wordt een conditioneel gemiddelde genoemd omdat het een gemiddelde uitkomst beschrijft, conditioneel op het feit dat \(X=x\).
Gezien
\[E[Y_i|X_i=x]=g(x)\]het gemiddelde beschrijft voor subjecten met een ESR1 expressieniveau van \(x\) is de foutterm \(\epsilon_i\) gemiddeld 0 voor deze subjecten:
\[E[\epsilon_i\vert X_i=x]=0.\]