Data exploratie en beschrijvende statistiek
Data exploratie is heel belangrijk om inzicht te krijgen in de data en is een essentiële eerste stap om te leren uit data. Het wordt vaak ondergewaardeerd of over het hoofd gezien.
Data in deze cursus wordt verwerkt via het statistisch software pakket R. Vooraleer we met de data exploratie van start kunnen gaan moeten we de data eerst importeren in R.
We geven duidelijk aan welke delen van de tekst over het Code gaan en welke over de interpretatie. Probeer initiëel te focussen op de interpretatie. Het Code zal je immers gedurende de eerste weken oppikken. Daarom hebben we ook gekozen om aparte clips te maken m.b.t interpretatie en Code. Zodat de manier hoe je de data analyse dient uit te voeren het begrip initiëel niet in de weg staan.
Importeer de data
Code Tijdens de data exploratie gaan we voor de data manipulaties
veelal gebruik maken van functies uit het tidyverse package. Dat kan
worden geladen door het commando library(tidyverse).
Via het commando read_lines kunnen we enkele regels van een data
bestand inlezen om de structuur van het data bestand te weten te komen.
library(tidyverse)
read_lines("https://raw.githubusercontent.com/statOmics/sbc20/master/data/armpit.csv")
## [1] "trt,rel" "placebo,54.99207606973059"
## [3] "placebo,31.84466019417476" "placebo,41.09947643979057"
## [5] "placebo,59.52063914780293" "placebo,63.573407202216075"
## [7] "placebo,41.48648648648649" "placebo,30.44041450777202"
## [9] "placebo,42.95676429567643" "placebo,41.7391304347826"
## [11] "placebo,33.896515311510036" "transplant,57.218124341412015"
## [13] "transplant,72.50900360144058" "transplant,61.89258312020461"
## [15] "transplant,56.690140845070424" "transplant,76"
## [17] "transplant,71.7357910906298" "transplant,57.757296466973884"
## [19] "transplant,65.1219512195122" "transplant,67.53424657534246"
## [21] "transplant,77.55359394703657"
We observeren de volgende structuur:
- Gegevens in het bestand zijn door comma’s gescheiden.
- Elke rij bevat de gegevens voor 1 proefpersoon
- Verschillende variabelen worden gemeten per persoon en zijn van elkaar gescheiden door een comma. Het bestand is in csv formaat: “comma separated values”.
- We kunnen bestanden met dit formaat inlezen R via het commando
read_csv. - We slaan de data op in R in het object met naam ap. Hiervoor
gebruiken we de
<-operator. - We geven de data tabel terug door het object aan te roepen door zijn naam te typen.
ap <- read_csv("https://raw.githubusercontent.com/statOmics/sbc20/master/data/armpit.csv")
ap
## # A tibble: 20 x 2
## trt rel
## <chr> <dbl>
## 1 placebo 55.0
## 2 placebo 31.8
## 3 placebo 41.1
## 4 placebo 59.5
## 5 placebo 63.6
## 6 placebo 41.5
## 7 placebo 30.4
## 8 placebo 43.0
## 9 placebo 41.7
## 10 placebo 33.9
## 11 transplant 57.2
## 12 transplant 72.5
## 13 transplant 61.9
## 14 transplant 56.7
## 15 transplant 76
## 16 transplant 71.7
## 17 transplant 57.8
## 18 transplant 65.1
## 19 transplant 67.5
## 20 transplant 77.6
Als we data matrix observeren zien we de volgende structuur:
proefpersonen in de rijen waarvoor we twee karateristieken (variabelen)
hebben bijgehouden per subject: behandeling (trt) en relatieve
abundantie (rel). Deze data structuur wordt tidy data genoemd.
Weinig mensen kunnen a.d.h.v. het bekijken van de data matrix structuur of patronen zien in de data. Daarom zullen we de data moeten verwerken en visualiseren.
Beschrijvende statistiek
In artikels en de media worden resultaten uit een steekproef vaak gerapporteerd a.d.h.v. gemiddelde en de standaardafwijking.
Code
- We vatten de data eerst samen. We berekenen het gemiddelde en de
standaard deviatie (een maat voor de spreiding, zie volgende
hoofdstukken). We slaan het resultaat hiervan op in het object
apRelSum via
apRelSum <-.
-
We pipen (via
%>%) hetapdataframe naar degroup_byfunctie om de data te groeperen per treatment trt:group_by(trt). -
We pipen het resultaat naar de
summarize_atfunction om de “rel” variable samen te vatten en berekenen hierbij het gemiddelde en standaardafwijking. Omdat we de data eerst hebben gegroepeerd zullen we het gemiddelde en de standaard deviatie berekenen per groep.
apRelSum <- ap %>%
group_by(trt) %>%
summarize_at("rel",
list(mean=mean,
sd=sd
)
)
We tonen vervolgens het resultaat door het object apRelSum aan te roepen
apRelSum
## # A tibble: 2 x 3
## trt mean sd
## <chr> <dbl> <dbl>
## 1 placebo 44.2 11.5
## 2 transplant 66.4 7.88
We kunnen ook een tabel in de webpagina of het pdf bestand integreren via het commando kable van het knitr pakket:
knitr::kable(apRelSum, 'html')
| trt | mean | sd |
|---|---|---|
| placebo | 44.15496 | 11.543251 |
| transplant | 66.40127 | 7.880175 |
Interpretatie
Het effect van de behandeling in de steekproef kan worden gekwantificeerd door de gemiddelde relatieve abundantie te vergelijken in elke steekproef. We observeren dat de gemiddelde relatieve abundantie in de steekproef gemiddeld 22.2% hoger is in de transplantatie dan in de placebo groep.
We quantificeren ook de variabiliteit of de spreiding van de gegevens rond het gemiddelde aan de hand van de standaardafwijking.
Het gemiddelde en de standaardafwijking wordt ook vaak grafisch weergegeven in een barplot.
Code We maken in deze cursus gebruik van het pakket ggplot2 om
grafieken te maken.
Met de ggplot2 bibliotheek kunnen we gemakkelijk grafieken opbouwen in
lagen (layers). Hierdoor leest de code veel makkelijker. In uitgebreide
introductie tot ggplot vinden je in de Dodona module R data exploration
and visualisation.
Bar plots worden heel veel gebruikt in artikels om resultaten weer te geven.
-
We pipen de samengevatte data naar de functie
ggplot. Dat is de basis van elke ggplot. We selecteren de variabele met de behandeling trt als x variabele en de variabele met naam mean als y-variabele voor de plot. We doen dit steeds via de aesteticsaesfunctie.aes(x=trt,y=mean) -
We maken een barplot door een laag toe te voegen via de
geom_barfunction. De statistiek isstat="identity"omdat de hoogte van de bar gelijk is aan de waarde voor y (hier het gemiddelde voor de relatieve abundantie). -
We voegen foutenvlaggen toe om de onzekerheid op het gemiddelde weer te geven. We doen dit via de
geom_errorbarfunctie en specifiëren het minimum en maximum van de error bar. Hetwidthargument wordt gebruikt om de breedte van de error bar smaller te maken dat deze van de bar.
apRelSum %>%
ggplot(aes(x=trt,y=mean)) +
geom_bar(stat="identity") +
geom_errorbar(aes(ymin=mean-sd,ymax=mean+sd),width=.2)
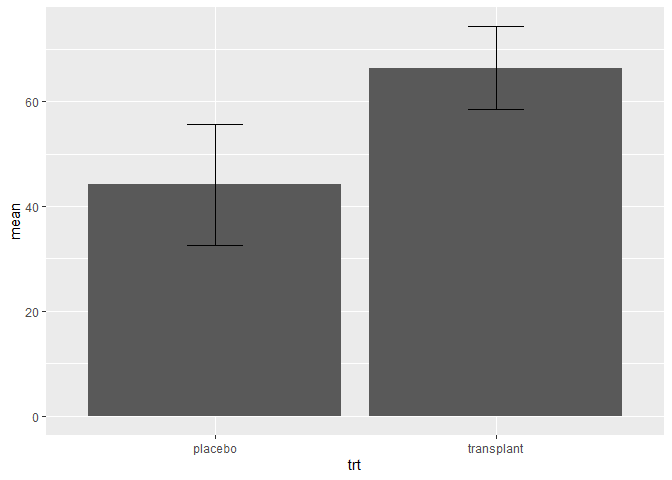
Interpretatie
Is deze plot informatief? Nee!
We zien enkel een “two-number summary”: het gemiddelde en de standaarddeviatie. We weten niet of deze wel een goeie samenvattende maat zijn voor de data. Bovendien wordt heel wat ruimte in de plot benut die niet informatief is: er zijn bijvoorbeeld geen observaties met relatieve abundanties van 0%.
Om de data van verschillende groepen makkelijk te kunnen vergelijken in een grafiek die meer informatief is dan de bar plot ontwikkelde Tukey de boxplot. Dit is een “five-number summary” het bereik van de data (minimum en maximum) samen met het 25, 50 (mediaan), and 75 percentiel. Deze percentielen zijn de waarden waarvoor respectievelijk 25%, 50% en 75% van de data in de steekproef kleiner zijn. Tukey raadde aan om het bereik van de data te berekenen zonder rekening te houden met outliers (extreme observaties). De outliers worden afzonderlijk als data punten toegevoegd aan de plot. In het hoofdstuk 4: Data Exploratie leggen we uit wat outliers precies zijn.
Code We maken nu een boxplot voor de ap data
- We pipen het
apdata object naarggplot - We selecteren de data voor de plot via
ggplot(aes(x=trt,y=rel)) - We voegen een laag toe voor de boxplot dmv de functie
geom_boxplot()
ap %>%
ggplot(aes(x=trt,y=rel)) +
geom_boxplot()
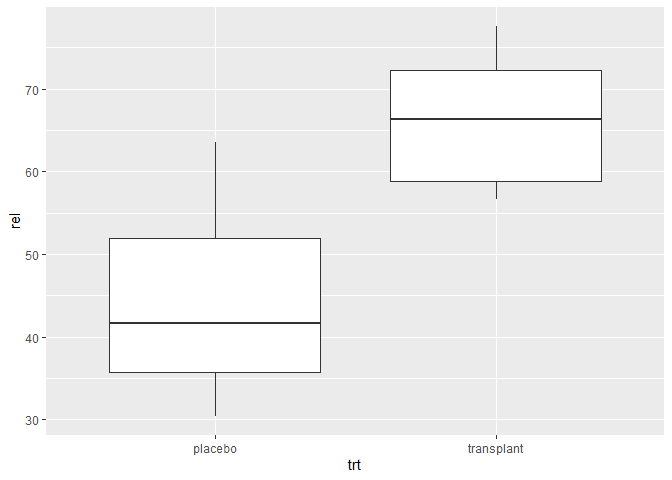
Interpretatie: De boxplot geeft ons al veel meer informatie dan de barplot. Het bereik van de data wordt weergegeven door de wiskers (eindpunten van de verticale lijnen in het midden van de boxplot). De box in de boxplot geeft het 25%, 50% en 75% percentiel weer. (Merk op dat er geen outliers zijn. Er worden geen individuele punten weergegeven in de plot)
We observeren dat boxplot voor de transplantie groep hoger ligt dan voor de placebo groep. Wat aangeeft dat de relatieve abundanties van Staphylococcus vaak hoger liggen voor personen in de steekproef die zijn toegewezen aan de transplantatie groep dan voor personen die zijn toegewezen aan placebo groep. Dit is een indicatie dat de behandeling werkt.
Verder weten we dat er maar 10 observaties zijn in elke groep. Dat laat toe om de ruwe gegevens aan de plot toe te voegen zonder dat de plot te druk wordt.
Code
- Merk op dat we het argument
outlier.shapeop NA (not available) zettenoutlier.shape=NAin thegeom_boxplotfunctie. Dat doen we standaard als we de ruwe data toevoegen aan de boxplots: anders zullen outliers immers twee keer worden weergegeven (eerst via de boxplot laag en daarna door de laag met alle ruwe data toevoegen aan de plot). - We geven de ruwe data weer via de
geom_point(position="jitter")functie. We gebruiken hierbij het argument position=‘jitter’ zodat we wat random ruis toevoegen aan de x-cordinaat zodat de gegevens elkaar niet overlappen.
ap %>%
ggplot(aes(x=trt,y=rel)) +
geom_boxplot(outlier.shape=NA) +
geom_point(position="jitter")

Dit is een informatieve plot! Het toont de data zo ruw mogelijk weer. De plot is toch nog goed leesbaar en toont duidelijk aan dat de relatieve abundanties bij bijna alle proefpersonen in de transplantatie groep hoger is dan deze voor de placebo groep.