In februari 2013 werden de broers Elwin en Yohan in Frankrijk gearresteerd voor een reeks van zes verkrachtingen. Beide ontkennen ze echter alle beschuldigingen. Uitmaken wie van de twee schuldig is, is echter niet zo eenvoudig — het zijn immers identieke tweelingen, waardoor de genetische verschillen tussen de twee miniem zijn. Politiechef Emmanual Kiehl van Marseille zei hierover: "Er zullen waarschijnlijk duizenden verschillende testen nodig zijn voor we kunnen beslissen wie van de twee de dader is."
Deze zaak is de zoveelste in een reeks juridische verwikkelingen waarbij DNA-materiaal wordt aangewend als bewijslast tegen identieke tweelingen. Tijdens een juwelenroof in Duitsland in januari 2009 lieten de dieven een druppel zweet achter op een latex handschoen. In een databank van misdadigers werden twee hits gevonden — identieke tweelingen Hassan en Abbas O. (de Duitse wet vereist geheimhouding van de familienaam van verdachten). Beide broers hebben een strafblad voor diefstal en fraude, maar toch werden ze alletwee vrijgelaten. De rechtbank oordeelde immers: "Uit de bewijslast die voorhanden is, kunnen we afleiden dat ten minste één van de twee broers bij de misdaad betrokken was, maar het was niet mogelijk om uit te maken wie van de twee."
Later dat jaar ontsnapte de identieke tweeling Sathis Raj en Sabarish Raj in Maleisië aan dood door ophanging, toen een rechter oordeelde dat het onmogelijk was om te bepalen wie van de twee schuldig was aan drugssmokkel. "Hoewel één van de twee zou moeten opgeroepen worden om zich te komen verdedigen, kan ik het niet maken om de verkeerde op te roepen", oordeelde de rechter. "Ik kan het ook niet maken om de verkeerde persoon naar de galg te sturen."
In 2003 had een vrouw uit Missouri binnen het tijdsbestek van enkele uren afzonderlijk sex met de identieke tweeling Raymon and Richard Miller. Toen ze zwanger bleek te zijn, ontkenden beide mannen de vader te zijn van het kind. In Missouri wordt een man enkel als de wettelijke vader erkend als een vaderschapstest een kans aangeeft van 98 procent of meer op een DNA-match. In dit geval toonde die voor de Miller-tweeling echter tweemaal een kans van meer dan 99.9 procent.
"Bij identieke tweelingen zou je zelfs geen verschil vinden als je hun volledige genoom zou sequeneren", vertelde de forensische wetenschapper Bob Gaensslen toentertijd aan ABC News. Recenter onderzoek toont echter aan dat dit niet het geval is, maar het uitpluizen van de verschillen kan wel een dure aangelegenheid zijn — in de zaak uit Marseille werd aan de politie verteld dat een dergelijke test €996,000 zou kosten.
En de reeks gaat verder. In Augustus 2013 probeerden de Briste autoriteiten uit te maken hoe ze iemand zouden kunnen vervolgen voor een verkrachting waarbij DNA-materiaal zowel Mohammed als Aftab Asghar had geidentificeerd. "Het is een ongewone zaak", zei officier van justitie Sandra Beck. "Het zijn identieke tweelingen. De beschuldiging is er één van verkrachting, maar er is bijkomend onderzoek nodig."
Opgave
De grootste verschillen in het genoom van identieke tweelingen zijn toe te schrijven aan copy-number variaties (CNV). Bij deze structurele variaties wordt het DNA van een genoom gewijzigd zodat cellen een abnormale of — voor bepaalde genen — een normale variatie hebben in het aantal herhalingen van één of meer DNA-fragmenten. CNVs corresponderen met relatief grote fragmenten van het genoom die verwijderd (deletie) of gedupliceerd (insertie) werden op bepaalde chromosomen. Zo kan een chromosoom dat normaal de fragmenten A-B-C-D heeft, in plaats daarvan de fragmenten A-B-C-C-C-D (een duplicatie van C) of A-B-D (een deletie van C) hebben.
Om CNVs op te sporen, veronderstellen we dat een DNA-sequentie is opgebouwd uit een prefix, gevolgd door een infix en een suffix, waarbij de infix bestaat uit $$n \in \mathbb{N}$$ herhalingen van een DNA-fragment dat we de kopie noemen.
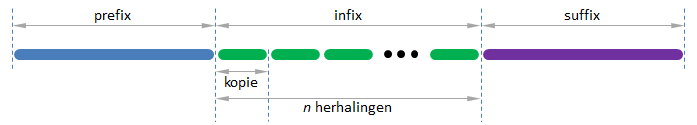
In deze opgave stellen we DNA-sequenties voor als strings die enkel bestaan uit de hoofdletters A, C, G en T. Veronderstel nu dat we beschikken over twee DNA-sequenties die enkel verschillen in het aantal herhalingen van de kopie. Dan kunnen we op basis van een vergelijking tussen de twee sequenties, de verschillende onderdelen identificeren waaruit ze zijn opgebouwd. Hiervoor gaan we als volgt te werk:
-
Schrijf een functie repliceer met één verplichte parameter kopie en drie optionele parameters aantal (standaardwaarde: 1), prefix (standaardwaarde: de lege string) en suffix (standaardwaarde: de lege string). Aan de parameter aantal moet een natuurlijk getal doorgegeven worden, en aan de andere parameters moet een DNA-sequentie doorgegeven worden. De functie moet de DNA-sequentie teruggeven die is opgebouwd uit de gegeven prefix, gevolgd door een infix en de gegeven suffix, waarbij de infix bestaat uit het opgegeven aantal herhalingen van de gegeven kopie.
-
Schrijf een functie kopie_aantal waaraan een DNA-sequentie $$s$$ moet doorgegeven worden. De functie moet een tuple bestaande uit een DNA-sequentie $$k$$ en een natuurlijk getal $$n \in \mathbb{N}_0$$ teruggeven, waarbij $$k$$ de kortst mogelijke DNA sequentie is waarvoor de gegeven DNA-sequentie $$s$$ bestaat uit $$n$$ herhalingen van $$k$$.
-
Schrijf een functie LGU waaraan twee DNA-sequenties moeten doorgegeven worden. De functie heeft ook nog een optionele parameter suffix (standaardwaarde: False) waaraan een Booleaanse waarde moet doorgegeven worden. Indien de waarde False wordt doorgegeven aan de parameter suffix, dan moet de functie de langste gemeenschappelijke prefix (langste gemeenschappelijke string aan het begin van de gegeven sequenties) teruggeven. Indien de waarde True wordt doorgegeven aan de parameter suffix, dan moet de functie de langste gemeenschappelijke suffix (langste gemeenschappelijke string aan het einde van de gegeven sequenties) teruggeven.
-
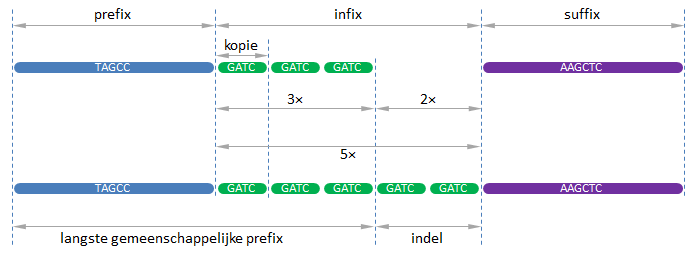
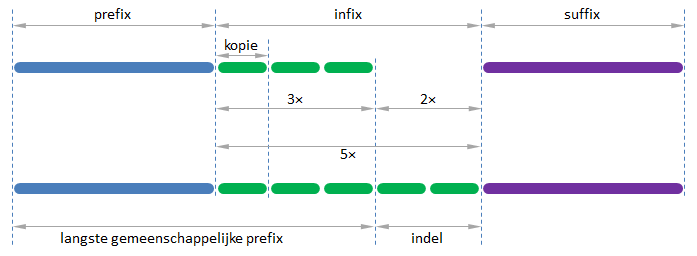
Gebruik nu de voorgaande twee functies om een functie CNV te schrijven waaraan twee DNA-sequenties moeten doorgegeven worden. Indien de twee DNA-sequenties eenzelfde prefix en suffix hebben, en enkel van elkaar verschillen in het aantal tussenliggende herhalingen van dezelfde kopie, dan moet de functie een tuple teruggeven dat bestaat uit de (kortst mogelijke) kopie en het aantal herhalingen van die kopie in de eerste en in de tweede sequentie. Anders moet de functie een AssertionError opwerpen met de boodschap geen CNV gevonden. Gebruik de volgende procedure om de verschillende onderdelen te identificeren waaruit de twee gegeven DNA-sequenties zijn opgebouwd (zie ook onderstaande figuur):
-
controleer dat de sequenties verschillend zijn, anders geen CNV gevonden
-
bepaal de langste gemeenschappelijk prefix (LGP) van de sequenties
-
bepaal de suffix als het resterende deel van de kortste sequentie (het deel dat niet behoort tot de LGP)
-
controleer dat de langste sequentie eindigt met de gevonden suffix, anders geen CNV gevonden
-
bepaal de indel als het deel tussen de LGP en de suffix in de langste sequentie
-
bepaal de kortst mogelijke kopie en het aantal herhalingen van die kopie waaruit de indel is opgebouwd (bij CNV bestaat de indel immers uit één of meer herhalingen van de kopie)
-
bepaal het aantal extra herhalingen van de kopie op het einde van de LGP
-
Voorbeeld
>>> repliceer('GATC')
'GATC'
>>> repliceer('GATC', aantal=4)
'GATCGATCGATCGATC'
>>> repliceer('GATC', aantal=2, prefix='TAGCC')
'TAGCCGATCGATC'
>>> repliceer(kopie='GATC', aantal=3, suffix='AAGCTC')
'GATCGATCGATCAAGCTC'
>>> repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC')
'TAGCCGATCGATCGATCAAGCTC'
>>> repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC')
'TAGCCGATCGATCGATCGATCGATCAAGCTC'
>>> kopie_aantal('CTCTCTCTCTCTCTCTCTCTCTCT') # repliceer(kopie='CT', aantal=12)
('CT', 12)
>>> kopie_aantal('GATCGATCGATCGATC') # repliceer(kopie='GATC', aantal=4)
('GATC', 4)
>>> kopie_aantal(repliceer('GATCGATC', aantal=2))
('GATC', 4)
>>> kopie_aantal('GATCGATCGATCGATCG') # repliceer(kopie='GATC', aantal=4) + 'G'
('GATCGATCGATCGATCG', 1)
>>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC')
>>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC')
>>> LGU(seq1, seq2)
'TAGCCGATCGATCGATC'
>>> LGU(seq1, seq2, suffix=True)
'CGATCGATCGATCAAGCTC'
>>> LGU(seq1, seq1)
'TAGCCGATCGATCGATCAAGCTC'
>>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC')
>>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC')
>>> CNV(seq1, seq2)
('GATC', 3, 5)
>>> CNV(seq2, seq1)
('GATC', 5, 3)
>>> CNV(seq1, seq1)
Traceback (most recent call last):
AssertionError: geen CNV gevonden
>>> seq3 = 'TAGCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, prefix='TAGC', suffix='AAGCTC')
>>> CNV(seq1, seq3)
('GATCGAT', 0, 1)
>>> seq4 = 'TAGCCGATCGATCGATCGATCGATCAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AGCTC', prefix='TAGCC')
>>> CNV(seq1, seq4)
Traceback (most recent call last):
AssertionError: geen CNV gevonden