Eiwitsequenties worden in deze opgave voorgesteld als strings die enkel hoofdletters bevatten. Elke hoofdletter stelt een aminozuur van de sequentie voor. Trypsine is een eiwitafbrekend enzym dat voedingseiwitten afbreekt in de dunne darm van de mens en verschillende diersoorten. Dit enzym heeft een zeer specifieke functie — het splitst alleen peptidebindingen waarvan de carboxylgroep afkomstig is van één van de basische aminozuren lysine (voorgesteld door de letter K) en arginine (voorgesteld door de letter R) — en wordt daarom in het laboratorium veel toegepast bij structureel onderzoek van eiwitten.
High-performance liquid chromatography (HPLC) is een scheidingstechniek die kan gecombineerd worden met shotgun tandem massaspectrometrische methoden om de actieve eiwitten in een biologisch staal te bepalen. Hierbij wordt een trypsine digest gebruikt om de eiwitten van het staal open te knippen in verschillende stukken na elke K of R in de sequentie. Deze afzonderlijke stukken worden tryptische peptiden genoemd. De sequentie van tryptische peptiden kan met een massaspectrometer bepaald worden. De meeste toestellen hebben echter een detectielimiet die enkel toelaat om peptiden met een lengte tussen 5 en 50 uit te lezen. Als de laatste peptide van de eiwitsequentie zelf niet op K of R eindigt, dan wordt ze ook niet opgepikt door de massaspectrometer.
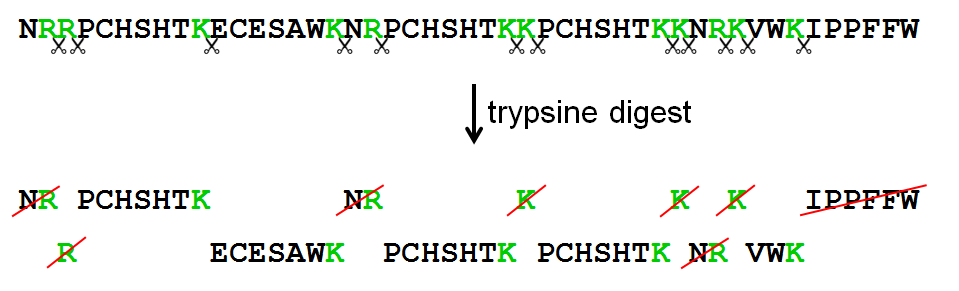
Toepassingen zoals Unipept bouwen een grote eiwitdatabank op, die tryptische peptiden bevat van meer dan 29 miljoen gekende eiwitten. Deze toepassing kan zowel de diversiteit als de functionele activiteit van een biologisch staal onderzoeken, door na te gaan welke eiwitten corresponderen met de tryptische peptiden die uit het staal gesequeneerd worden.
Opgave
Definieer een klasse eiwitDB waarmee eenvoudige eiwitdatabanken kunnen aangemaakt worden. In deze eiwitdatabanken kan dan gezocht worden naar eiwitten die een opgegeven lijst van tryptische peptiden bevatten. De objecten van de klasse eiwitDB moeten de volgende methoden hebben:
Een initialisatiemethode die ervoor zorgt dat elk nieuw aangemaakt object van de klasse eiwitDB een eigenschap peptiden heeft die verwijst naar een dictionary. Bij een nieuw aangemaakt object is deze dictionary nog leeg, maar gaandeweg kan deze dictionary opgevuld worden met strings als sleutels en verzamelingen van strings als waarden.
Een methode peptideToevoegen die kan gebruikt worden om een nieuwe tryptische peptide aan de databank toe te voegen. Aan deze methode moeten twee argumenten doorgegeven worden: een string die het label van een eiwitsequentie bevat, en een string die een tryptische peptide bevat. Enkel tryptische peptiden met lengte tussen 5 en 50 (grenzen inbegrepen) mogen aan de databank toegevoegd worden. Bovendien moet een tryptische peptide eindigen op K of R, en mag elders in de sequentie geen K of R voorkomen. De methode moet een AssertionError met de tekst ongeldige peptide opwerpen als er een peptide wordt doorgegeven die niet aan deze voorwaarden voldoet. Het toevoegen van een tryptische peptide aan de databank gebeurt door in de dictionary waarnaar de eigenschap peptiden verwijst, het label van de eiwitsequentie toe te voegen aan de verzameling die correspondeert met de sleutel die gelijk is aan de peptide die aan de methode werd doorgegeven. Als de dictionary nog geen sleutel heeft voor de gegeven peptide, dan moet er in de dictionary een nieuw sleutel-waarde paar toegevoegd worden, met de peptide als sleutel en als waarde een verzameling die enkel het gegeven label bevat.
Een methode eiwitToevoegen die kan gebruikt worden om alle tryptische peptiden met lengte tussen 5 en 50 (grenzen inbegrepen) van een gegeven eiwitsequentie aan de databank toe te voegen. Aan deze methode moeten twee argumenten doorgegeven worden: een string die het label van de eiwitsequentie bevat, en een string met de eiwitsequentie zelf. De methode moet de eiwitsequentie opbreken in tryptische peptiden, en elk van deze peptiden aan de databank toevoegen onder het label van de eiwitsequentie.
Een methode eiwittenToevoegen waaraan een bestandsnaam als argument moet doorgegeven worden. Deze bestandsnaam moet verwijzen naar een tekstbestand, waarvan elke regel een label en een eiwitsequentie bevat, van elkaar gescheiden door een tab. Deze methode moet alle peptiden van alle eiwitten uit het bestand toevoegen aan de databank.
Een methode identificeer die kan gebruikt worden om eiwitten uit de databank te identificeren. Aan deze methode moet een collectieobject (dat kan dus een lijst, tuple, verzameling, … zijn) doorgegeven worden, waarin een aantal peptiden vervat zitten. De methode moet een alfabetisch gesorteerde lijst teruggeven met de labels van alle eiwitten uit de databank waarin elk van de gegeven peptiden minstens één keer voorkomt.
Zorg er bij de implementatie van al deze methoden voor dat je optimaal gebruik maakt van de methoden die je reeds eerder geïmplementeerd hebt.
Voorbeeld
In onderstaande voorbeeldsessie gaan we ervan uit dat het bestand eiwitten.txt zich in de huidige directory bevindt.
>>> unipept = eiwitDB()
>>> unipept.peptideToevoegen('PROT0001', 'ECESAWK')
>>> unipept.peptiden
{'ECESAWK': {'PROT0001'}}
>>> unipept.peptideToevoegen('PROT0002', 'WHK')
Traceback (most recent call last):
AssertionError: ongeldige peptide
>>> unipept.peptideToevoegen('PROT0002', 'ESHLSTLAVQENEIG')
Traceback (most recent call last):
AssertionError: ongeldige peptide
>>> unipept.peptideToevoegen('PROT0002', 'NWAQNAKIGGADWDCVCR')
Traceback (most recent call last):
AssertionError: ongeldige peptide
>>> unipept.eiwitToevoegen('PROT0002', 'HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY')
>>> unipept.peptiden
{'ECESAWK': {'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}}
>>> unipept.eiwitToevoegen('PROT0003', 'NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW')
>>> unipept.peptiden
{'ECESAWK': {'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}}
>>> unipept.eiwitToevoegen('PROT0004', 'YEMWQHPGEQHKECESAWKVPYCGFITRPCHSHTKECESAWK')
>>> unipept.peptiden
{'ECESAWK': {'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'VPYCGFITR': {'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0004', 'PROT0002'}}
>>> unipept.identificeer(['VPYCGFITR'])
['PROT0004']
>>> unipept.identificeer({'ECESAWK', 'PCHSHTK'})
['PROT0002', 'PROT0003', 'PROT0004']
>>> unipept.identificeer(('YEMWQHPGEQHK', 'ECESAWK', 'PCHSHTK'))
['PROT0002', 'PROT0004']
>>> unipept.identificeer({'PCHSHTK', 'VPYCGFITR'})
['PROT0004']
>>> unipept.eiwittenToevoegen('eiwitten.txt')
>>> unipept.peptiden
{'ECESAWK': {'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'VCEFPWFPMLINDVCR': {'PROT0007'}, 'VPYCGFITR': {'PROT0005', 'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0002'}, 'PCHSHTK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'CSFHCLEK': {'PROT0006'}, 'AFNYMMPNTK': {'PROT0006'}, 'AYDDEVASFPGCMMATK': {'PROT0007', 'PROT0006'}, 'FIPYYPIYSR': {'PROT0006'}, 'TLCHETMR': {'PROT0005'}, 'HTPNYGVMWMFMNEWMSYDR': {'PROT0006', 'PROT0005'}, 'CDQMHVFDIYMIAIACSWGGPPSLTK': {'PROT0007'}, 'FGHSMTR': {'PROT0005'}}
>>> unipept.identificeer(('YEMWQHPGEQHK', 'VPYCGFITR', 'ECESAWK'))
['PROT0004', 'PROT0005']
>>> unipept.identificeer(('PCHSHTK', 'AYDDEVASFPGCMMATK'))
['PROT0006']
>>> unipept.identificeer(['NEGNLNVMK'])
[]