Röntgendiffractie is een techniek die gebruikt wordt om de structuur van vaste stoffen te bepalen. Als de techniek toegepast wordt op kristallen, dan kan daarmee de kristalstructuur zeer nauwkeurig bepaald worden. Uit het diffractiepatroon — de verstrooiing van de röntgenstralen — is het immers mogelijk om de positie van de atomen in de moleculen te reconstrueren. In de vroege jaren '50 begonnen wetenschappers deze techniek toe te passen op biologische moleculen zoals DNA en eiwitten. De techniek wordt ook gebruikt voor het bepalen van de structuur van mineralen, eenvoudige en meer complexe organische verbindingen.

Röntgendiffractie is een zeer nauwkeurige microscopische techniek. Waar atoomkrachtmicroscopie op dit ogenblik maar net toelaat om individuele atomen te zien, kan de positie van de atomen met röntgendiffractie nog 100-1000 maal nauwkeuriger bepaald worden.
Opgave
We modelleren de verstrooiing van röntgenstralen die aanbotsen tegen de atomen van een vlakke molecule, door het rechthoekig gebied dat wordt ingenomen door de molecule onder te verdelen in $$m$$ rijen en $$n$$ kolommen ($$m, n \in \mathbb{N}_0$$). Elk atoom van de molecule ligt in een cel van dit rechthoekig rooster, en cellen bevatten nooit meer dan één atoom.
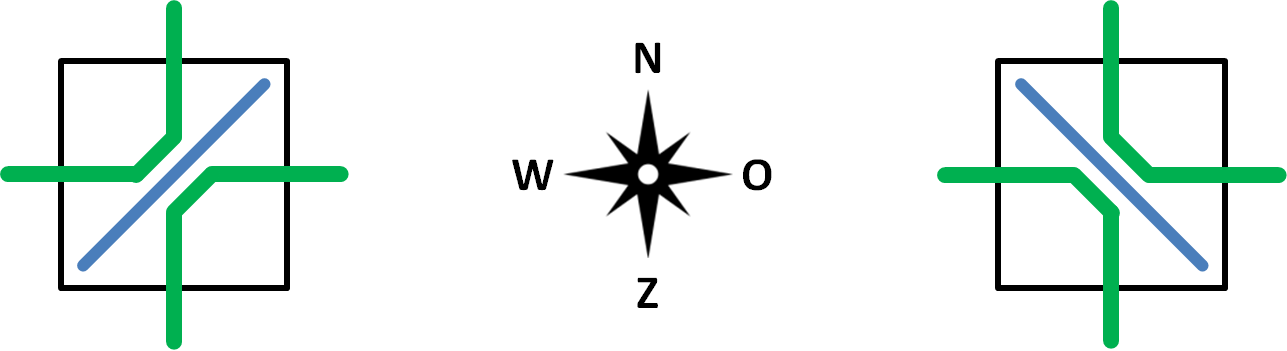
De röntgenstralen die we laten invallen op de molecule zijn steeds gericht naar het noorden, oosten, zuiden of westen. Op basis van de manier waarop ze invallende röntgenstralen afbuigen worden atomen onderverdeeld in twee categorieën. Atomen met voorwaartse reflectie (links in onderstaande figuur) buigen röntgenstralen die naar het oosten gericht zijn af naar het noorden (en vice versa) en buigen röntgenstralen die naar het zuiden gericht zijn af naar het westen (en vice versa). Atomen met een achterwaartse reflectie (rechts in onderstaande figuur) buigen röntgenstralen die naar het oosten gericht zijn af naar het zuiden (en vice versa) en buigen röntgenstralen die naar het noorden gericht zijn af naar het westen vice versa).
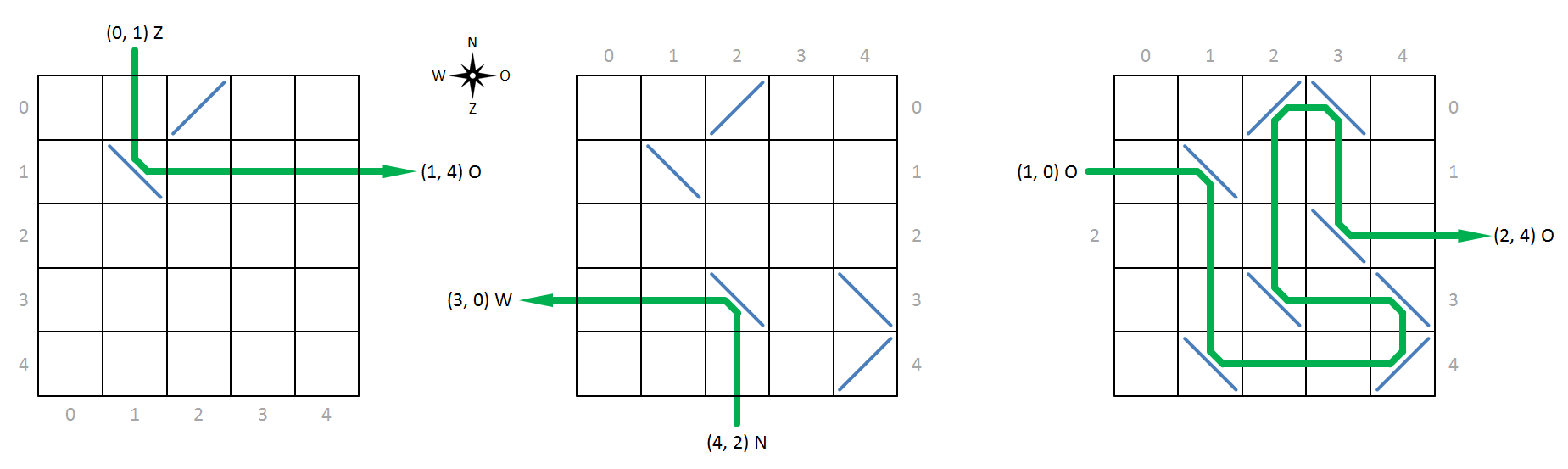
Om te kunnen verwijzen naar specifieke cellen in het rooster, nummeren we de rijen oplopend van noord naar zuid en de kolommen van west naar oost, waarbij we steeds beginnen te nummeren vanaf nul (grijze getallen op onderstaande figuur). We kunnen de positie van een cel in het rooster dan voorstellen door een tuple $$(r, k)$$, waarbij $$r$$ het nummer van de rij en $$k$$ het nummer van de kolom is waarop de cel gelegen is. Een röntgenstraal die invalt op een molecule beschrijven we aan de hand van de positie van de cel waar de straal op de molecule invalt, samen met de richting waarnaar de straal gericht is. Je opdracht bestaat erin om voor een invallende röntgenstraal te bepalen waar en in welke richting hij de molecule zal verlaten.
Hieronder tonen we enkele voorbeelden van röntgenstralen die invallen op een molecule en door de atomen afgebogen worden. De linkse molecule heeft bijvoorbeeld een atoom met voorwaartse reflectie op positie (0, 2) en een atoom met achterwaartse reflectie op positie (1, 1). Zoals aangegegeven door de groene lijn zal een röntgenstraal die naar het zuiden gericht is en invalt op positie (0, 1) door het atoom op positie (1, 1) afgebogen worden naar het oosten en de molecule in die richting verlaten op positie (1, 4).
Gevraagd wordt om een klasse Molecule te schrijven waarvan elk object een molecule voorstelt in de vorm van een rechthoekig $$m \times n$$ rooster. Elke cel van het rooster kan een atoom bevatten met voorwaartse of achterwaartse reflectie, dat zich gedraagt zoals hierboven beschreven. De klasse moet minstens de volgende methoden ondersteunen:
-
Een initialisatiemethode waaraan het aantal rijen $$m \in \mathbb{N_0}$$ en het aantal kolommen $$n \in \mathbb{N_0}$$ van het rooster moet doorgegeven worden. Na initialisatie van een object bevinden er zich nog geen atomen op het rooster, maar tijdens de levensduur van het object kunnen atomen met voorwaartse of achterwaartse reflectie op het rooster geplaatst worden door het aanroepen van de methoden atoom en atomen.
-
Een methode atoom waarmee een atoom op het rooster kan geplaatst worden. Aan de methode moet de positie doorgegeven worden van de cel waarop het atoom moet geplaatst worden. Indien de gegeven positie buiten het rooster ligt of indien op deze positie reeds een atoom geplaatst werd, dan moet de methode een AssertionError opwerpen met de boodschap ongeldige positie. Standaard heeft het te plaatsen atoom een voorwaartse reflectie. De methode heeft echter ook nog een tweede optionele parameter voorwaarts waaraan een Booleaanse waarde kan doorgegeven worden (standaardwaarde: True). Indien aan deze parameter de waarde False doorgegeven wordt, dan heeft het te plaatsen atoom een achterwaartse reflectie.
-
Een methode atomen waarmee één of meer atomen met dezelfde soort reflectie op het rooster kunnen geplaatst worden. De posities van deze atomen moeten als een collectie (een lijst, tuple of verzameling) aan de methode doorgegeven worden. Deze posities moeten aan dezelfde voorwaarden voldoen als de positie die wordt doorgegeven aan de methode atoom. Indien dit niet het geval is, dan moet de methode een AssertionError opwerpen met de boodschap ongeldige positie. De methode heeft ook nog een tweede optionele parameter voorwaarts die op dezelfde manier moet geïnterpreteerd worden als bij de methode atoom, en die het soort reflectie aangeeft van de atomen die op het rooster moeten geplaatst worden.
-
Een methode __str__ die een stringvoorstelling teruggeeft van de atomen die reeds op het rooster geplaatst werden. Deze stringvoorstelling bestaat uit $$m$$ regels, die elk $$n$$ symbolen bevatten die telkens van elkaar gescheiden worden door een spatie. Het $$k$$-de symbool op de $$r$$-de regel (regels en symbolen genummerd vanaf nul) geeft aan of de cel op positie $$(r, k)$$ een atoom bevat, en welk soort reflectie dat atoom heeft. Atomen met voorwaartse reflectie worden voorgesteld door een slash (/) en atomen met achterwaartse reflectie door een backslash (\). Cellen zonder atoom worden aangeduid met een punt (.).
-
Een methode afbuiging waaraan twee argumenten moeten doorgegeven worden, die de coördinaten van een invallende röntgenstraal voorstellen: i) de positie van de cel waar de straal op de molecule invalt en ii) de richting waarnaar de straal gericht is. Deze richting wordt aangeduid met één van de volgende letters: N voor noord, Z voor zuid, W voor west en O voor oost. De methode moet een lijst van posities in het rooster teruggeven die de volgorde aangeeft van de cellen waarlangs de röntgenstraal passeert bij zijn doortocht doorheen de molecule. Hierbij wordt de straal door de atomen afgebogen zoals beschreven in de inleiding van de opgave.
-
Een methode diffractie waaraan dezelfde twee argumenten (met dezelfde betekenis) moeten doorgegeven worden als aan de methode afbuiging. De methode moet een tuple teruggeven dat twee elementen bevat: i) de positie van de cel waar de invallende röntgenstraal de molecule terug verlaat, en ii) de richting waarin de straal de molecule verlaat.
Opmerking: Strings in Python gebruiken het backslash karakter als escape symbool. Daarom moet een letterlijke backslash in een string verdubbeld worden: '\\'.
Voorbeeld
Merk op dat een docstring een gewone string is, waardoor elke letterlijke backslash die voorkomt in de docstring moet verdubbeld worden. Klik hier om onderstaande interactieve sessie om te vormen tot een docstring.
>>> molecule = Molecule(5, 5)
>>> molecule.atoom((0, 2))
>>> molecule.atoom((1, 1), voorwaarts=False)
>>> print(molecule)
. . / . .
. \ . . .
. . . . .
. . . . .
. . . . .
>>> molecule.afbuiging((0, 1), 'Z')
[(0, 1), (1, 1), (1, 2), (1, 3), (1, 4)]
>>> molecule.diffractie((0, 1), 'Z')
((1, 4), 'O')
>>> molecule.atoom((4, 4))
>>> molecule.atomen([(3, 2), (3, 4)], voorwaarts=False)
>>> print(molecule)
. . / . .
. \ . . .
. . . . .
. . \ . \
. . . . /
>>> molecule.afbuiging((4, 2), 'N')
[(4, 2), (3, 2), (3, 1), (3, 0)]
>>> molecule.diffractie((4, 2), 'N')
((3, 0), 'W')
>>> molecule.atomen({(0, 3), (2, 3), (4, 1)}, voorwaarts=False)
>>> print(molecule)
. . / \ .
. \ . . .
. . . \ .
. . \ . \
. \ . . /
>>> molecule.afbuiging((1, 0), 'O')
[(1, 0), (1, 1), (2, 1), (3, 1), (4, 1), (4, 2), (4, 3), (4, 4), (3, 4), (3, 3), (3, 2), (2, 2), (1, 2), (0, 2), (0, 3), (1, 3), (2, 3), (2, 4)]
>>> molecule.diffractie((1, 0), 'O')
((2, 4), 'O')
>>> molecule.atoom((6, 6))
Traceback (most recent call last):
AssertionError: ongeldige positie
>>> molecule.atoom((1, 1))
Traceback (most recent call last):
AssertionError: ongeldige positie