Het FASTQ formaat wordt gebruikt om zowel DNA-sequenties als de daarmee geassocieerde kwaliteitsscores op te slaan in een tekstbestand. Het formaat werd oorspronkelijk ontwikkeld door het Wellcome Trust Sanger Institute, maar is ondertussen zowat de de facto standaard geworden om next-generation sequencing resultaten op te slaan.
Een FASTQ bestand bevat een reeks sequentierecords, waarbij elke record bestaat uit vier opeenvolgende regels:
-
regel 1 bevat een apestaartje (@) gevolgd door een sequence identifier en een optionele beschrijving
-
regel 2 bevat een reeks sequentieletters
-
regel 3 bevat een plusteken (+), optioneel gevolgd door dezelfde sequence identifier en eventueel een bijkomende beschrijving
-
regel 4 bevat de corresponderende kwaliteitsscore voor elke sequentieletter uit regel 2
Omwille van de eenvoud worden in dit formaat zowel de sequentieletters als de kwaliteitsscores voorgesteld door één enkel ASCII karakter. Bij wijze van voorbeeld ziet de inhoud van het FASTQ bestand sample.fq met zes sequentierecords er als volgt uit:
@sample sequence 1
GGAAGTCCATGGAGTTATTTGCGGTAACCGGGACCGGCCT
+sample sequence 1
ba^aaabaa`]baaaaa_aab]D^^`b`aYDW]abaa`^a
@sample sequence 2
CTCCTTATAGCATCATACGCACAGCTGGTATCCCCATTGCTTATTAGGGTTCAGCCCTGTCGCTCTCCCC
+sample sequence 2
ababa``aaaababaaaabaa_``aa``]^aa_`aa_Z_aaaaa^baaaaaaaaaaaaaaaaaa`^```a
@sample sequence 3
GGAAGTCCATGGAGTTATTTGCGGTAACCGGGACCGGCCTGGGGTTAGATTTTATGTCGT
+sample sequence 3
ba^aaabaa`]baaaaa_aab]D^^`b`aYDW]abaa`^ababbaaabaaaaa`]`ba`]
@sample sequence 4
TATAC
+sample sequence 4
baab`
@sample sequence 5
CTCCTTATAGCATCATACGCACAGCTGGTATCCCCATTGCTTATTAGGGT
+sample sequence 5
ababa``aaaababaaaabaa_``aa``]^aa_`aa_Z_aaaaa^baaaa
@sample sequence 6
GGAAGTCCATGGAGTTATTTGCGGTAACCGGGACCGGCCTGGGGTTAGATTTTATGTCGT
+sample sequence 6
ba^aaabaa`]baaaaa_aab]D^^`b`aYDW]abaa`^ababbaaabaaaaa`]`ba`]Voorafgaand aan het sequeneren zelf, wordt eerst een genomische bibliotheek opgebouwd. Als een artefact van de PCR amplificatie die daarmee gepaard gaat, kan het gebeuren dat eenzelfde fragment meerdere keren wordt uitgelezen. Daarom worden de DNA-sequenties in een FASTQ bestand doorgaans eerst ontdubbeld, om een mogelijke bias te vermijden bij het analyseren van de sequenties. Bij het ontdubbelen worden niet de volledige sequenties in rekening gebracht, maar wordt enkel gekeken naar een prefix die bestaat uit de eerste $$n$$ basen van de sequentie.
Als we bijvoorbeeld prefixen van lengte 10 beschouwen, dan zien we in het voorbeeld FASTQ bestand dat de records 1, 3, en 6 dezelfde prefix GGAAGTCCAT hebben, en dat ook de records 2 en 4 dezelfde prefix CTCCTTATAG hebben. We hebben de records uit dit FASTQ bestand hieronder grafisch voorgesteld, waarbij we dezelfde prefixen ook dezelfde kleur gegeven hebben.
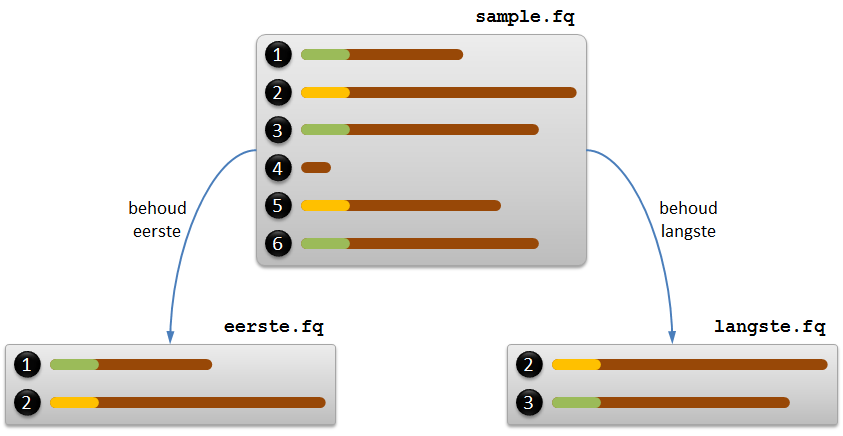
Voor het ontdubbelen van de sequenties in een FASTQ bestand worden in de praktijk twee strategieën gebruikt. Beide strategieën hebben gemeenschappelijk dat alle records waarvan de sequentie korter is dan $$n$$ (de lengte van de prefix) altijd verwijderd worden. Als $$n = 10$$, dan is dit bijvoorbeeld het geval voor record 4 in het voorbeeld FASTQ bestand.
Bij de eerste strategie — die het linkse resultaat uit bovenstaande figuur oplevert — wordt een FASTQ record enkel behouden als we de prefix van de sequentie voor het eerst te zien krijgen. Voor de implementatie van deze strategie volstaat het om de FASTQ records één voor één te overlopen en een verzameling bij te houden van de prefixen die reeds gezien werden. Een FASTQ record wordt dan enkel behouden als de prefix nog niet in de verzameling voorkomt.
Bij de tweede strategie — die het rechtse resultaat uit bovenstaande figuur oplevert — wordt voor elke prefix enkel de record met de langste sequentie behouden. Voor de implementatie van deze strategie moeten de records van het originele FASTQ bestand nu twee keer overlopen worden. De eerste keer worden de records overlopen om een dictionary op te bouwen die elke prefix van sequenties langer dan $$n$$ afbeeldt op de lengte van de langste sequentie met die prefix. De tweede keer worden de records opnieuw één voor één overlopen, en worden enkel de records behouden waarvan de lengte van de sequentie correspondeert met de lengte waarop de prefix van de sequentie wordt afgebeeld door de dictionary. Indien er voor eenzelfde prefix meerdere records zijn waarvoor de lengte van de sequentie correspondeert met de lengte waarop de prefix van de sequentie wordt afgebeeld door de dictionary, dan moet enkel de eerste record daarvan behouden worden. In het voorbeeld FASTQ bestand hebben de sequenties van records 3 en 6 bijvoorbeeld dezelfde prefix en dezelfde lengte (de maximale lengte van alle records met die prefix), en moet dus enkel record 3 behouden worden. De sequentie van record 1 heeft ook dezelfde prefix, maar die sequentie is korter dan die van records 3 en 6.
Opgave
In deze opgave werken we enkel met FASTQ bestanden die DNA-sequenties bevatten. DNA-sequenties worden hierbij voorgesteld als strings die enkel bestaan uit de hoofdletters A, C, G en T. Gevraagd wordt:
-
Schrijf een functie behoud_eerste waaraan drie argumenten moeten doorgegeven worden: i) een string met de locatie van een FASTQ bestand waarvan de records moeten ontdubbeld worden, ii) een string met de locatie van een nieuw aan te maken FASTQ bestand waar de behouden records naartoe moeten weggeschreven worden in dezelfde volgorde waarin ze voorkomen in het gegeven FASTQ bestand, en iii) de lengte $$n \in \mathbb{N}_0$$ van de prefixen die moeten gebruikt worden bij het ontdubbelen. Voor het ontdubbelen moet de eerste strategie geïmplementeerd worden die in de inleiding van de opgave staat beschreven.
-
Schrijf een functie vind_langste waaraan twee argumenten moeten doorgegeven worden: i) een string met de locatie van een FASTQ bestand en ii) een getal $$n \in \mathbb{N}_0$$. De functie moet een dictionary teruggeven, die elke prefix van lengte $$n$$ die voorkomt in het gegeven FASTQ bestand (beperkt tot prefixen van sequenties die minstens lengte $$n$$ hebben) afbeeldt op de maximale lengte van de sequenties met die prefix.
-
Gebruik de functie vind_langste om een functie behoud_langste te schrijven. Aan deze functie moeten dezelfde drie argumenten doorgegeven worden als bij de functie behoud_eerste, met dezelfde betekenis. Voor het ontdubbelen moet nu evenwel de tweede strategie geïmplementeerd worden die in de inleiding van de opgave staat beschreven.
In de praktijk bevatten FASTQ bestanden de resultaten van next-generation sequencing experimenten, die bestaan uit enkele miljoenen reads (korte DNA-sequenties). Dergelijke bestanden zijn te groot om volledig in het computergeheugen te passen. Zorg er bij de implementatie van de ontdubbelingsfuncties daarom voor dat niet de volledige inhoud van het gegeven FASTQ bestand in het geheugen wordt geladen, maar dat enkel geheugen gebruikt wordt voor de informatie van één FASTQ record, samen met een verzameling (functie behoud_eerste) of en dictionary (functie behoud_langste) met prefixen die gebruikt wordt om te beslissen of die FASTQ record al dan niet moet uitgeschreven worden.
Voorbeeld
Bij onderstaande voorbeeldsessie gaan we ervan uit dat het FASTQ bestand sample.fq zich in de huidige directory bevindt. Dit bestand heeft dezelfde inhoud als het voorbeeldbestand uit de inleiding van deze opgave.
>>> behoud_eerste('sample.fq', 'eerste.fq', 10)
>>> print(open('eerste.fq', 'r').read().rstrip())
@sample sequence 1
GGAAGTCCATGGAGTTATTTGCGGTAACCGGGACCGGCCT
+sample sequence 1
ba^aaabaa`]baaaaa_aab]D^^`b`aYDW]abaa`^a
@sample sequence 2
CTCCTTATAGCATCATACGCACAGCTGGTATCCCCATTGCTTATTAGGGTTCAGCCCTGTCGCTCTCCCC
+sample sequence 2
ababa``aaaababaaaabaa_``aa``]^aa_`aa_Z_aaaaa^baaaaaaaaaaaaaaaaaa`^```a
>>> vind_langste('sample.fq', 10)
{'GGAAGTCCAT': 60, 'CTCCTTATAG': 70}
>>> behoud_langste('sample.fq', 'langste.fq', 10)
>>> print(open('langste.fq', 'r').read().rstrip())
@sample sequence 2
CTCCTTATAGCATCATACGCACAGCTGGTATCCCCATTGCTTATTAGGGTTCAGCCCTGTCGCTCTCCCC
+sample sequence 2
ababa``aaaababaaaabaa_``aa``]^aa_`aa_Z_aaaaa^baaaaaaaaaaaaaaaaaa`^```a
@sample sequence 3
GGAAGTCCATGGAGTTATTTGCGGTAACCGGGACCGGCCTGGGGTTAGATTTTATGTCGT
+sample sequence 3
ba^aaabaa`]baaaaa_aab]D^^`b`aYDW]abaa`^ababbaaabaaaaa`]`ba`]