Als de DNA sequentie van een organisme bepaald is dan stelt zich de vraag welke gebieden van het genoom coderen voor eiwitten. Om te voorspellen waar deze coderende gebieden precies liggen, probeert men de eiwitten uit te lezen in de zes reading frames. Hierbij is een reading frame niets anders dan een manier om de sequentie van nucleotiden in een DNA molecule op te delen in een reeks opeenvolgende, niet-overlappende tripletten — zo een reeks van drie nucleotiden wordt in deze context een codon genoemd.
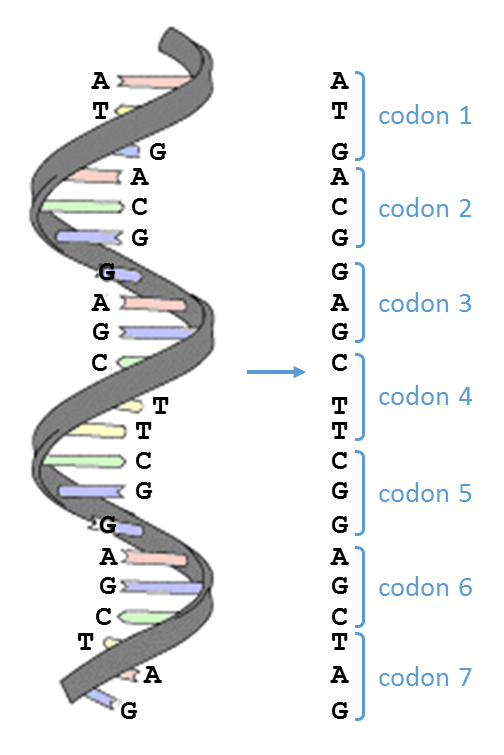
Bij de vertaling van DNA naar eiwitten spelen bepaalde codons een bijzondere rol:
-
startcodon (ATG): hier kan de vertaling van een DNA sequentie naar een eiwit beginnen
-
stopcodon (TAG, TGA, TAA): hier eindigt de vertaling van een DNA sequentie naar een eiwit
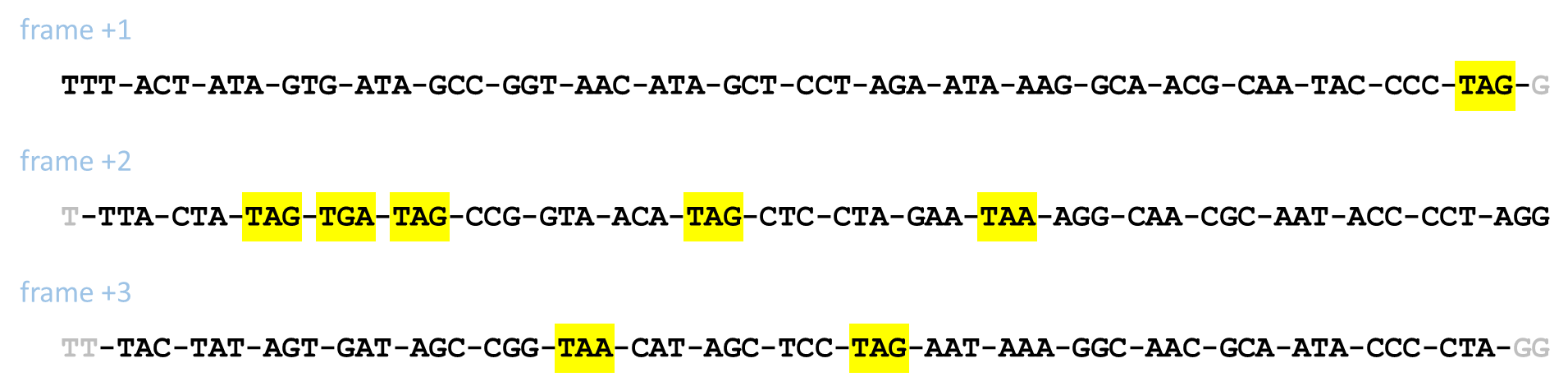
Bij het omzetten van DNA naar eiwitten kan de vertaling echter op elke mogelijke positie van de DNA sequentie beginnen. Het eerste codon kan dus ofwel bestaan uit de eerste drie nucleotiden van de DNA sequentie, of we kunnen één of twee nucleotiden overslaan en dan bestaat het eerste codon telkens uit de volgende drie nucleotiden. Als gevolg daarvan kan een DNA sequentie op drie mogelijke manieren opgedeeld worden in codons, die we frame +1, frame +2 en frame +3 noemen. Beschouw bij wijze van voorbeeld de DNA sequentie
TTTACTATAGTGATAGCCGGTAACATAGCTCCTAGAATAAAGGCAACGCAATACCCCTAGG
Deze sequentie kan op de volgende drie manieren opgedeeld worden in
codons. Hierbij hebben we de stopcodons in het geel gemarkeerd.
DNA is echter dubbelstrengig en elk van deze strengen kan dienen als de coderende streng voor eiwitten. Gelukkig kunnen we voor een gegeven DNA sequentie makkelijk de sequentie van de andere streng bepalen als diens invers complement. Het invers complement van een DNA sequentie wordt verkregen door de string om te keren en het complement te nemen van elke base (A en T zijn complementaire basen, net zoals C en G). Naast het nemen van het complement moeten we de string ook omkeren omwille van de gerichtheid van DNA. DNA-replicatie en -transcriptie gebeuren immers van het 5' uiteinde tot het 3' uiteinde, en het 3' uiteinde van de ene streng ligt tegenover het 5' uiteinde van de complementaire streng. Als we dus gewoon het complement zouden nemen, dan zou de tweede streng in de verkeerde richting gelezen worden.

Daarom zijn er in totaal niet drie maar zes reading frames: drie in de gegeven DNA sequentie en drie in het invers complement van de gegeven DNA sequentie. Het invers complement wordt op dezelfde drie manieren opgedeeld in codons als de originele sequentie, maar nu worden deze opdelingen respectievelijk frame -1, frame -2 en frame -3 genoemd. Het invers complement van de voorbeeldsequentie is
CCTAGGGGTATTGCGTTGCCTTTATTCTAGGAGCTATGTTACCGGCTATCACTATAGTAAA
en deze kan op de volgende manier opgedeeld worden in nog eens drie frames, waarbij we opnieuw de stopcodons in het geel aangeduid hebben.

Omdat er geen stopcodons mogen voorkomen in een reading frame dat codeert voor een eiwit, vormt het detecteren van stopcodons dus een eerste belangrijke stap bij het bepalen van de gebieden van een genoom die coderen voor eiwitten. Aangezien er bij de voorbeeldsequentie geen stopcodons voorkomen in reading frame -2, is dit dus vermoedelijk het reading frame dat kan gebruikt worden om het eiwit te vertalen.
Opgave
Bepaal de zes reading frames van een gegeven DNA sequentie en tel hoeveel stopcodons er in elk reading frame voorkomen. Hierbij stellen we DNA sequenties voor als strings die enkel bestaan uit de letters A, C, G en T (zowel in hoofdletters als in kleine letters). Gevraagd wordt:
-
Schrijf een functie isStopcodon waaraan een DNA sequentie moet doorgegeven worden. De functie moet een Booleaanse waarde teruggeven, die aangeeft of de gegeven DNA sequentie een stopcodon is.
-
Schrijf een functie inversComplement waaraan een DNA sequentie moet doorgegeven worden. De functie moet het invers complement van de gegeven DNA sequentie teruggeven in hoofdletters.
-
Schrijf een functie stopcodons waaraan twee argumenten moeten doorgegeven worden: een DNA sequentie en het nummer van een reading frame (+1, +2, +3, -1, -2 of -3). De functie moet het aantal stopcodons teruggeven die voorkomen in het gegeven reading frame van de gegeven DNA sequentie.
-
Schrijf een functie codons waaraan twee argumenten moeten doorgegeven worden: een DNA sequentie en het nummer van een reading frame (+1, +2, +3, -1, -2 of -3). De functie moet een stringvoorstelling teruggeven van de opdeling in codons van de gegeven DNA sequentie in het gegeven reading frame. Hierbij moeten de codons en de fragmenten van één of twee nucleotiden vooraan en achteraan de sequentie telkens van elkaar gescheiden worden door een koppelteken (-).
Opmerking: In Python kan je voor een positieve integer optioneel ook een plusteken plaatsen (in technische termen is dit de unary plus operator1) om expliciet aan te geven dat het getal positief is. Maar los daarvan stellen +42 en 42 exact dezelfde waarde voor.
Voorbeeld
>>> isStopcodon('TAA')
True
>>> isStopcodon('tag')
True
>>> isStopcodon('ATC')
False
>>> inversComplement('AAGTC')
'GACTT'
>>> inversComplement('agcttcgt')
'ACGAAGCT'
>>> inversComplement('AGTCTTACGCTTA')
'TAAGCGTAAGACT'
>>> seq = 'TTTACTATAGTGATAGCCGGTAACATAGCTCCTAGAATAAAGGCAACGCAATACCCCTAGG'
>>> stopcodons(seq, +1)
1
>>> stopcodons(seq, +2)
5
>>> stopcodons(seq, +3)
2
>>> stopcodons(seq, -1)
3
>>> stopcodons(seq, -2)
0
>>> stopcodons(seq, -3)
1
>>> codons(seq, +1)
'TTT-ACT-ATA-GTG-ATA-GCC-GGT-AAC-ATA-GCT-CCT-AGA-ATA-AAG-GCA-ACG-CAA-TAC-CCC-TAG-G'
>>> codons(seq, +2)
'T-TTA-CTA-TAG-TGA-TAG-CCG-GTA-ACA-TAG-CTC-CTA-GAA-TAA-AGG-CAA-CGC-AAT-ACC-CCT-AGG'
>>> codons(seq, +3)
'TT-TAC-TAT-AGT-GAT-AGC-CGG-TAA-CAT-AGC-TCC-TAG-AAT-AAA-GGC-AAC-GCA-ATA-CCC-CTA-GG'
>>> codons(seq, -1)
'CCT-AGG-GGT-ATT-GCG-TTG-CCT-TTA-TTC-TAG-GAG-CTA-TGT-TAC-CGG-CTA-TCA-CTA-TAG-TAA-A'
>>> codons(seq, -2)
'C-CTA-GGG-GTA-TTG-CGT-TGC-CTT-TAT-TCT-AGG-AGC-TAT-GTT-ACC-GGC-TAT-CAC-TAT-AGT-AAA'
>>> codons(seq, -3)
'CC-TAG-GGG-TAT-TGC-GTT-GCC-TTT-ATT-CTA-GGA-GCT-ATG-TTA-CCG-GCT-ATC-ACT-ATA-GTA-AA'