Sangersequenering is een methode die gebruikt wordt om het DNA van een organisme te bepalen. De methode werd in 1977 ontwikkeld door Frederick Sanger en zijn collega's, en was gedurende 25 jaar de meest gebruikte sequeneringsmethode. Bij de Sangermethode worden de vier basen waaruit het DNA is opgebouwd (voorgesteld door de letters A, C, G en T) radioactief of fluorescent gelabeld, zodat opeenvolgende basen achtereenvolgens kunnen uitgelezen worden. Dit uitlezen gebeurt aan de hand van vier afzonderlijke detectoren, die voor elke positie nagaan of er een radioactief of fluorescent signaal gemeten wordt voor één van de vier mogelijke basen. Dit levert dus vier afzonderlijke signalen op die daarna kunnen samengevoegd worden tot één enkel signaal, waaruit de eigenlijke DNA sequentie kan samengesteld worden.
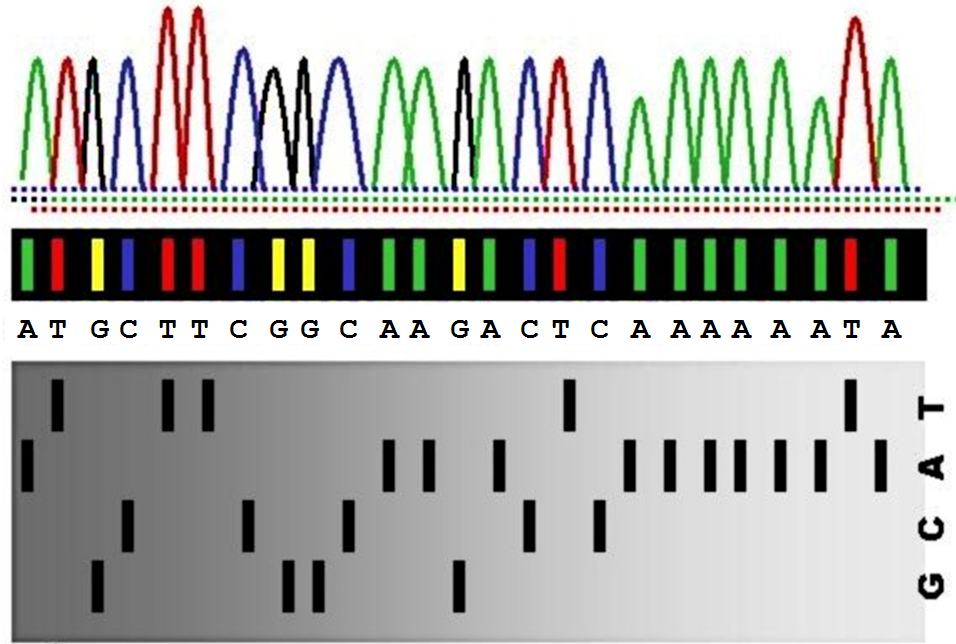
Als de detector voor base X (waarbij X staat voor één van de letters A, C, G of T) op een bepaalde positie een signaal heeft opgepikt, dan wordt dit voorgesteld aan de hand van de letter X. Als er geen signaal werd opgepikt, dan wordt dit voorgesteld met een koppelteken (-). Op die manier worden de signalen van de vier afzonderlijke detectoren — zoals die gemeten worden voor de DNA sequentie ATGCTTCGGCAAGACTCAAAAAATA — als volgt voorgesteld:
1111111111222222
1234567890123456789012345
detector A: A---------AA-A---AAAAAA-A
detector C: ---C--C--C----C-C--------
detector G: --G----GG---G------------
detector T: -T--TT---------T-------T-
=========================
DNA sequentie: ATGCTTCGGCAAGACTCAAAAAATA
Opgave
Schrijf een functie sanger waaraan een string als argument moet doorgegeven worden die enkel bestaat uit de hoofdletters A, C, G en T. Dit stringargument stelt een DNA sequentie voor. De functie moet als resultaat een string teruggeven waarin de gegeven DNA sequentie op de volgende manier wordt opgemaakt:
Centraal staan vier regels die de afzonderlijke signalen weergeven zoals die worden opgepikt door de detectoren voor de basen A, C, G en T. Deze regels bevatten dus enkel koppeltekens en baseletters op de posities waar een signaal werd opgepikt.
Onder deze vier regels staat een regel die evenveel gelijkheidstekens (=) bevat als er basen zijn in de gegeven DNA sequentie.
Helemaal onderaan staat de gegeven DNA sequentie zelf.
Boven de regels met de afzonderlijke signalen staan er nog een aantal regels die de posities van de basen binnen de DNA sequentie omschrijven. Posities worden hierbij geïndexeerd vanaf 1. Eerst komt een regel met de cijfers van de eenheden, daarboven een regel met de cijfers van de tientallen, daarboven een regel met de cijfers van de honderdtallen, enzoverder. Als er bijvoorbeeld geen tientallen moeten weergegeven worden, dan moet daarvoor ook geen regel uitgeschreven worden (analoog voor de honderdtallen, duizendtallen, …). Voor een positie die geen cijfer heeft voor de tientallen (voorloopnullen worden niet uitgeschreven) moet een spatie uitgeschreven worden op de regel voor de tientallen (analoog voor de honderdtallen, duizendtallen, …).
De laatste regel van de string die door de functie sanger teruggegeven wordt, mag niet eindigen op een newline. Bekijk onderstaande interactieve Python sessie voor enkele voorbeelden van hoe de opmaak moet gebeuren.
Voorbeeld
>>> print(sanger('ATGCTTCGG'))
123456789
A--------
---C--C--
--G----GG
-T--TT---
=========
ATGCTTCGG
>>> print(sanger('ATGCTTCGGCAAGACTCAAAAAATA'))
1111111111222222
1234567890123456789012345
A---------AA-A---AAAAAA-A
---C--C--C----C-C--------
--G----GG---G------------
-T--TT---------T-------T-
=========================
ATGCTTCGGCAAGACTCAAAAAATA