Het genoom van de meeste bacteriën bestaat uit één enkele circulaire DNA-molecule. DNA-replicatie — het proces waarbij DNA verdubbeld wordt — start op een bepaalde plaats in het genoom die in het Engels de origin of replication (kortweg ori) genoemd wordt, en wordt vanaf die plaats in twee richtingen tegelijkertijd uitgevoerd (zie onderstaande figuur). Het verdubbelingsproces eindigt op de plaats in het genoom die in het Engels de terminus of replication genoemd wordt.
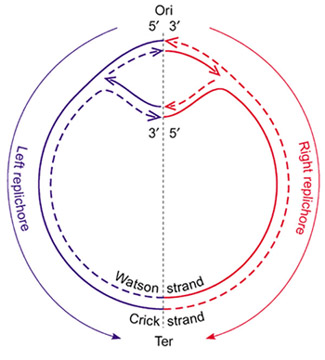
De specifieke structuur van de origin en terminus of replication verschilt van soort tot soort, maar er zijn enkele gemeenschappelijke eigenschappen zoals een hoge graad van AT (adenine en thymine zijn makkelijker van elkaar te scheiden omdat ze slechts twee waterstofbindingen vormen, terwijl guanine en cytosine er drie vormen). De origin of replication bindt ook met het prereplicatiecomplex, een eiwitcomplex dat DNA herkent, afwikkelt en begint te kopiëren.
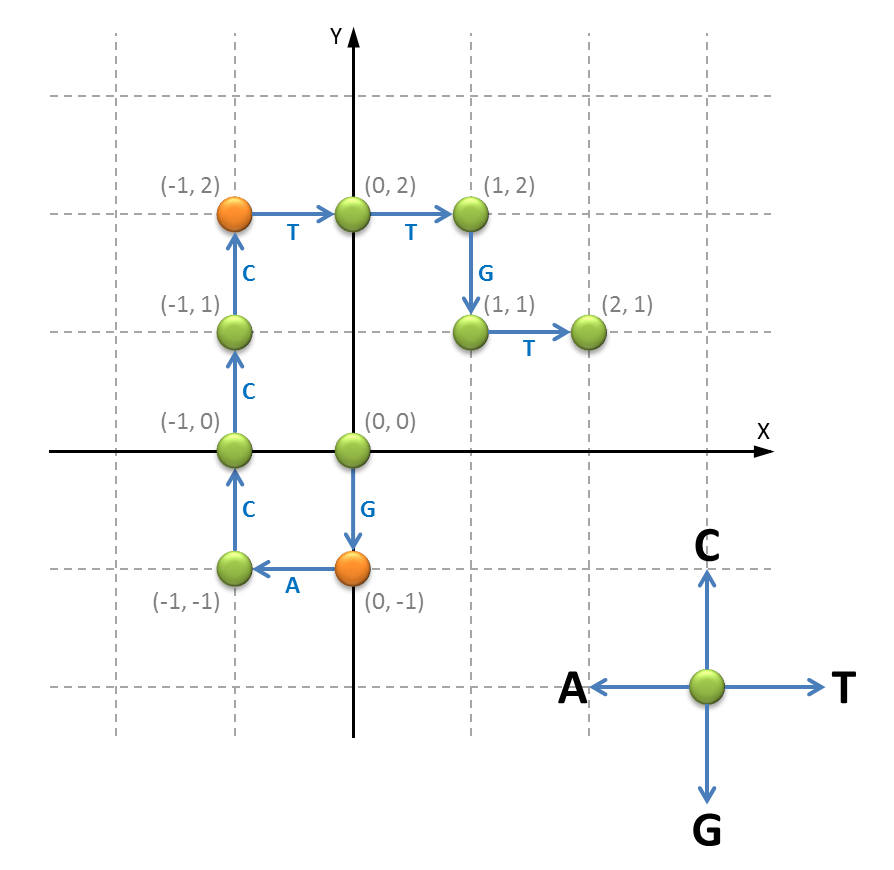
Een eenvoudige manier om de positie van de origin en terminus of replication in een genoomsequentie te vinden, maakt gebruik van een vectorvoorstelling van de DNA-molecule. Deze vector wordt opgebouwd als een lijst (list) van $$(x, y)$$-coördinaten (tuple; $$x, y \in \mathbb{N}$$ (int)) die start bij het punt $$(0, 0)$$ in de oorsprong. Voor elke opeenvolgende base in de DNA-sequentie wordt dan naar een buurpunt gesprongen: voor base A naar de linkerbuur, voor base T naar de rechterbuur, voor base C naar de bovenbuur en voor base G naar de onderbuur. De buren liggen telkens op afstand één van het vorige punt. Hieronder zie je bijvoorbeeld de vectorvoorstelling van de DNA-sequentie GACCCTTGT grafisch weergegeven.
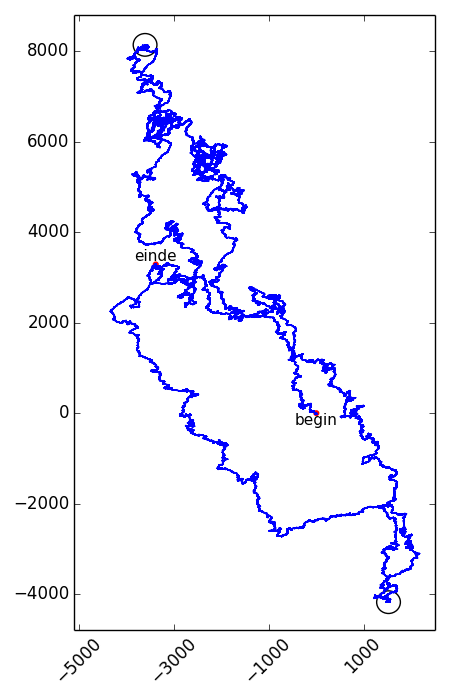
De posities in de vectorvoorstelling waar de $$y$$-coördinaat voor het eerst zijn maximale en minimale waarde bereikt, corresponderen met de posities van de origin en terminus op het genoom. Er kan echter niet eenduidig vastgelegd worden welke van de twee posities de origin en welke de terminus is. Hieronder zie je bijvoorbeeld de vectorvoorstelling van de volledige genoomsequentie van Haemophilus influenzae stam Rd (L420231), waarbij de posities van de origin en terminus of replication worden aangegeven met zwarte cirkels.
Opgave
We stellen DNA-sequenties voor als strings (str) die enkel bestaan uit de hoofdletters A, C, G en T. Gevraagd wordt:
-
Schrijf een functie vector waaraan een DNA-sequentie (str) moet doorgegeven worden. De functie moet de vectorvoorstelling van de DNA-sequentie teruggeven.
-
Schrijf een functie replicatie waaraan een DNA-sequentie (str) moet doorgegeven worden. De functie moet een tuple (tuple) van twee gehele getallen (int) teruggeven die de posities in de vectorvoorstelling van de DNA-sequentie aangeven waar de $$y$$-coordinaat voor het eerst zijn maximale — resp. minimale — waarde bereikt. De posities in de vectorvoorstelling worden oplopend genummerd vanaf nul.
-
Schrijf een functie sequentie waaraan de vectorvoorstelling van een DNA-sequentie moet doorgegeven worden. De functie moet de DNA-sequentie (str) teruggeven die met de vectorvoorstelling correspondeert.
Voorbeeld
>>> vector('GACCCTTGT')
[(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)]
>>> vector('CTGGGGTAA')
[(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)]
>>> replicatie('GACCCTTGT')
(5, 1)
>>> replicatie('CTGGGGTAA')
(1, 6)
>>> sequentie([(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)])
'GACCCTTGT'
>>> sequentie([(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)])
'CTGGGGTAA'Bronnen
-
Lobry JR (1996). A simple vectorial representation of DNA sequences for the detection of replication origins in bacteria. Biochimie 78, 323-326. 3
-
Mackiewicz P, Mackiewicz D, Kowalczuk M, Cebrat S (2001). Flip-flop around the origin and terminus of replication in prokaryotic genomes. Genome Biology 2(12). 4