Het genoom van een organisme is het geheel aan erfelijke informatie in een cel. Deze erfelijke informatie zit ofwel gecodeerd in DNA of — voor sommige soorten virussen — in RNA. Genen zijn structurele componenten van DNA en RNA die coderen voor een polypeptide of voor een RNA keten die een bepaalde functie vertolkt voor het organisme. Onderstaande afbeelding toont bijvoorbeeld de verschillende genen die gelegen zijn op het ringvormig mitochondriaal DNA (mtDNA) van een mens. De pijlen die gebruikt worden bij de voorstelling van de genen, geven aan dat ze zowel voorwaarts als achterwaarts kunnen georiënteerd zijn (DNA is dubbelstrengig). Een gen wordt gelocaliseerd op een genoom door de positie van de eerste en de laatste base van het gen op te geven. Posities worden aangegeven ten opzichte van de start van het genoom (bij ringvormige genomen wordt een startpositie gekozen), die zelf index 1 krijgt.
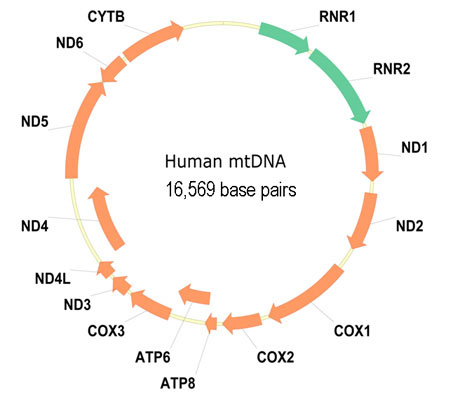
Wat uit bovenstaande afbeelding minder duidelijk is, is dat genen elkaar ook (gedeeltelijk) kunnen overlappen. Dat is duidelijker te zien bij de structurele opbouw van het HIV virus (een RNA lentivirus dat AIDS veroorzaakt) dat hieronder wordt weergegeven. De genen worden hierbij als rechthoeken aangeduid (RNA heeft immers maar één streng, waardoor alle genen in dit geval voorwaarts georiënteerd zijn).
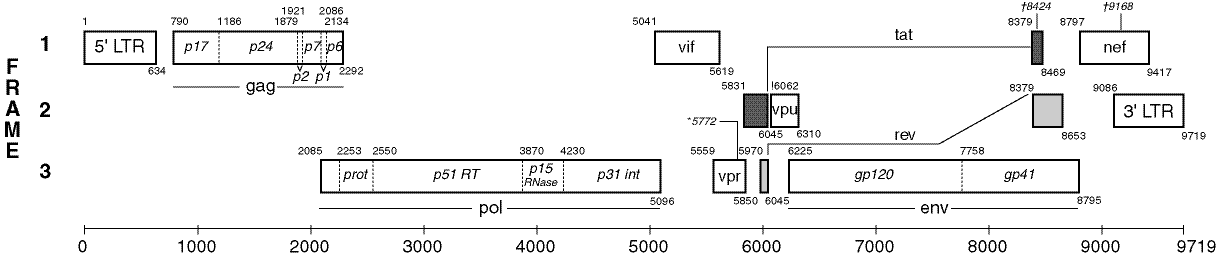
De dichtheid van een genoom is een percentage dat aangeeft hoeveel posities binnen het genoom bedekt zijn door genen (of andere structurele elementen). De genoomdichtheid kan bepaald worden door de lengtes van de genen te sommeren, en dit te delen door de lengte van het genoom. Dit is echter een naïeve manier van werken, die geen rekening houdt met overlappende genen en dus sommige posities meerdere keren in rekening kan brengen. Een betere manier bestaat erin te bepalen wat het percentage is van de posities binnen het genoom die binnen minstens één gen gelegen zijn.
Opgave
Definieer een klasse Gen waarmee genobjecten kunnen voorgesteld worden die over volgende methoden beschikken:
Een initialisatiemethode waaraan een startpositie en een stoppositie als argument moeten doorgegeven worden. De startpositie geeft aan waar de eerste base van het gen gelegen is, en de stoppositie waar de laatste base gelegen is. Als de stoppositie kleiner is dan de startpositie, dan is het gen achterwaarts georiënteerd.
Een methode __len__ die de lengte van het gen als resultaat teruggeeft.
Een methode __repr__ die de stringvoorstelling van een gen teruggeeft onder de vorm "Gen(start, stop)", waarbij start en stop respectievelijk de start- en stopposities van het gen voorstellen.
Een methode __str__ die de stringvoorstelling van een gen teruggeeft onder de vorm "start..stop" voor voorwaartse genen, en onder de vorm "complement(stop..start)" voor achterwaartse genen. Hierbij stellen start en stop respectievelijk de start- en stopposities van het gen voor.
Definieer ook nog een klasse Genoom waarmee genoomobjecten kunnen voorgesteld worden. Genoomobjecten moeten kunnen bijhouden waar hun genen gelegen zijn, en beschikken over volgende methoden:
Een initialisatiemethode waaraan de lengte van het genoom als argument moet doorgegeven worden.
Een methode __len__ die de lengte van het genoom als resultaat teruggeeft.
Een methode genToevoegen waaraan een genobject als argument moet doorgegeven worden. Door deze methode (verschillende keren) aan te roepen, kan opgegeven worden waar de verschillende genen op het genoom gelegen zijn. De methode moet een AssertionError met de tekst ongeldige coordinaat opwerpen indien de start- of stoppositie van het gegeven genobject niet binnen de grenzen van het genoom ligt.
Een methode dichtheid die de genoomdichtheid als een floating point waarde teruggeeft. Deze methode heeft een optionele Booleaans parameter overlap met als standaardwaarde True. Als de waarde False als argument wordt doorgegeven aan de parameter overlap, dan moet de genoomdichtheid op de naïeve manier berekend worden, zonder rekening te houden met mogelijke overlappende genen. Als de waarde True als argument wordt doorgegeven aan de parameter overlap, dan moet de genoomdichtheid berekend worden rekening houdend met mogelijke overlappende genen.
Voorbeeld
>>> gen1 = Gen(3309, 4264)
>>> len(gen1)
956
>>> gen1
Gen(3309, 4264)
>>> print(gen1)
3309..4264
>>> gen2 = Gen(14675, 14151)
>>> len(gen2)
525
>>> gen2
Gen(14675, 14151)
>>> print(gen2)
complement(14151..14675)
>>> hiv = Genoom(9719)
>>> len(hiv)
9719
>>> hiv.genToevoegen(Gen(1, 634))
>>> hiv.genToevoegen(Gen(790, 2292))
>>> hiv.genToevoegen(Gen(2085, 5096))
>>> hiv.genToevoegen(Gen(5041, 5619))
>>> hiv.genToevoegen(Gen(5559, 5850))
>>> hiv.genToevoegen(Gen(5831, 6045))
>>> hiv.genToevoegen(Gen(5970, 6045))
>>> hiv.genToevoegen(Gen(6062, 6310))
>>> hiv.genToevoegen(Gen(6225, 8795))
>>> hiv.genToevoegen(Gen(8379, 8424))
>>> hiv.genToevoegen(Gen(8379, 8653))
>>> hiv.genToevoegen(Gen(8797, 9417))
>>> hiv.genToevoegen(Gen(9086, 9719))
>>> hiv.dichtheid()
98.2302706039716
>>> hiv.dichtheid(overlap=False)
110.16565490276777
>>> hiv.genToevoegen(Gen(8888, 9999))
Traceback (most recent call last):
AssertionError: ongeldige coordinaat