In 2020 ontwikkelden Pfizer en BioNTech een vaccin tegen het SARS-CoV-2 virus. Het bevat genetisch materiaal dat sterk lijkt op dat van het beruchte Spike-eiwit van het virus: het eiwit dat zorgt voor de karakteristieke knotsvormige uitsteeksels aan de buitenkant van coronavirussen.
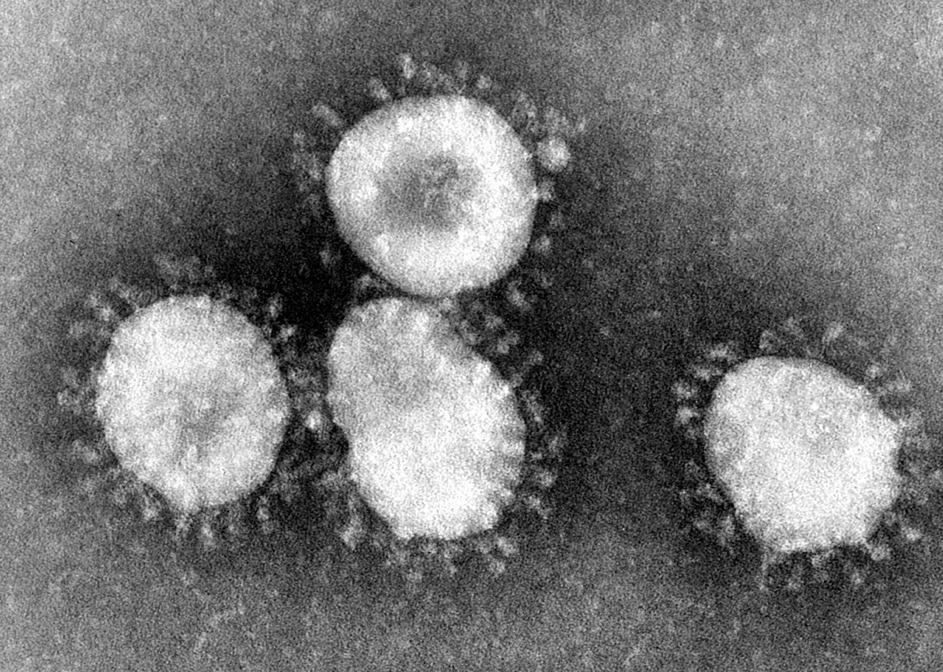
Via een slimme chemische processen zorgt het vaccin ervoor dat dit genetische materiaal in bepaalde cellen van het menselijk lichaam terechtkomt. Deze cellen beginnen dan plichtsgetrouw grote hoeveelheden SARS-CoV-2 Spike-eiwitten te produceren. Wanneer ons immuunsysteem deze Spike-eiwitten waarneemt, ontwikkelt het een krachtige reactie tegen het Spike-eiwit en het productieproces ervan.
Opgave
Een peptide is een kort stukje genetisch materiaal dat opgebouwd is uit een aantal opeenvolgende bouwstenen. Elke bouwsteen wordt voorgesteld door één karakter (str), waardoor peptiden als strings (str) kunnen voorgesteld worden. Verschillende karakters stellen verschillende soorten bouwstenen voor. Er wordt geen onderscheid gemaakt tussen hoofdletters en kleine letters.
Bij DNA en RNA zijn er vier soorten bouwstenen (basen). DNA-basen worden voorgesteld door de letters A, C, G en T. Bij RNA wordt de base T vervangen door de base U. Daardoor weten we meteen hoe DNA in RNA kan omgezet worden (en omgekeerd). Bij eiwitten zijn er 21 soorten bouwstenen (aminozuren) die voorgesteld worden door 20 letters en een sterretje (*; technisch gezien geen aminozuur maar een stopcodon).
Een DNA- of RNA-peptide kan beschrijven hoe een eiwit moet aangemaakt worden. Daarbij wordt de peptide opgedeeld in codons: een reeks van drie opeenvolgende basen. Er zijn dus $$4^3 = 64$$ verschillende codons. Omdat elk codon correspondeert met één aminozuur, moeten er dus meerdere codons zijn die met hetzelfde aminozuur corresponderen. Dergelijke codons worden synoniemen genoemd.
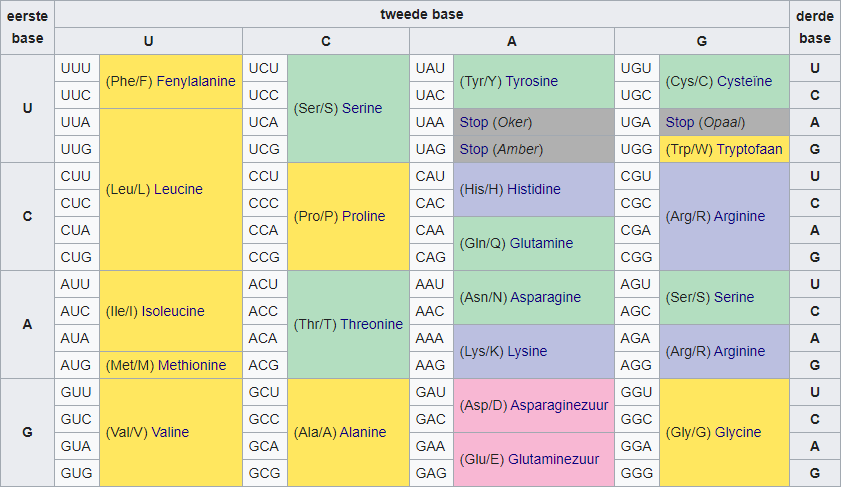
Een genetische code beschrijft hoe codons omgezet worden naar aminozuren. Er worden verschillende genetische codes (met lichte afwijkingen) gebruikt door verschillende groepen organismen. Een genetische code wordt in het volgende formaat opgeslagen in een tekstbestand. De eerste regel is een hoofding (en mag dus genegeerd worden). Daarna volgen 64 regels die elk de omzetting van een codon naar een aminozuur beschrijven met vier informatievelden die van elkaar gescheiden worden door komma's: i) codon, ii) naam van aminozuur, iii) drieletterige afkorting van aminozuur en iv) éénletterige afkorting van aminozuur. Dit is bijvoorbeeld hoe de standaard genetische code opgeslagen wordt:
Codon,Full Name,Abbreviation (3 Letter),Abbreviation (1 Letter) TTT,Phenylalanine,Phe,F TTC,Phenylalanine,Phe,F TTA,Leucine,Leu,L … TAA,Termination (ochre),Ter,* TAG,Termination (amber),Ter,* … GGC,Glycine,Gly,G GGA,Glycine,Gly,G GGG,Glycine,Gly,G
Een bestand met de beschrijving van een genetische code kan ofwel DNA-codons of RNA-codons bevatten. De codons kunnen zowel in hoofdletters als in kleine letters geschreven zijn.
Het Pfizer/BioNTech-vaccin maakt gebruik van een techniek die codonoptimalisatie genoemd wordt. Daarbij wordt elk codon van een peptide vervangen door een synoniem met zoveel mogelijk C's en G's. Daardoor wordt nog steeds hetzelfde eiwit beschreven, maar RNA met meer C's en G's1 wordt blijkbaar efficiënter omgezet naar eiwitten2. Dit zien we bijvoorbeeld reeds als we het begin van het Spike-eiwit van het SARS-CoV-2 virus vergelijken met het begin van het vaccin:
M F V F L V L L P L V S S Q C V
virus: AUG UUU GUU UUU CUU GUU UUA UUG CCA CUA GUC UCU AGU CAG UGU GUU
| | | | | | | | | | | | |
vaccin: AUG UUC GUG UUC CUG GUG CUG CUG CCU CUG GUG UCC AGC CAG UGU GUU
M F V F L V L L P L V S S Q C V
Alle mutaties zijn synoniemen en op één na introduceren ze allemaal een extra C of G in het RNA van het vaccin. Zo krijgen we dus een vaccin dat 95% effectief is. Gevraagd wordt:
-
Schrijf een functie genetische_code waaraan de locatie (str) van een tekstbestand met de beschrijving van een genetische code $$\mathcal{C}$$ moet doorgegeven worden. De functie moet een dictionary (dict) teruggeven die elk DNA-codon (str; in hoofdletters) afbeeldt op het corresponderende aminozuur (str; éénletterige afkorting; in hoofdletters). Dit wordt de dictionary-voorstelling van genetische code $$\mathcal{C}$$ genoemd.
Opmerking
Een genetische code kan in een tekstbestand zowel met DNA-codons als met RNA-codons beschreven worden, en die kunnen zowel in hoofdletters als in kleine letters staan. De dictionary-voorstelling van een genetische code gebruikt echter altijd DNA-codons in hoofdletters, en ook de letters voor de aminozuren staan altijd in hoofdletters.
-
Schrijf een functie omgekeerde_genetische_code waaraan de dictionary-voorstelling van een genetische code $$\mathcal{C}$$ moet doorgegeven worden. De functie moet een dictionary (dict) teruggeven die elk aminozuur (str) afbeeldt op een verzameling (set) met alle synonieme DNA-codons (str) die met dit aminozuur corresponderen volgens genetische code $$\mathcal{C}$$.
-
Schrijf een functie synoniemen waaraan twee argumenten moeten doorgegeven worden: i) een codon $$c$$ (str; DNA of RNA; hoofdletters of kleine letters) en ii) de dictionary-voorstelling van een genetische code $$\mathcal{C}$$. De functie moet een verzameling (set) teruggeven met alle synoniemen (str; in hoofdletters) van codon $$c$$ volgens genetische code $$\mathcal{C}$$ (inclusief het codon zelf). De functie heeft ook nog een optionele parameter RNA waaraan een Booleaanse waarde (bool) kan doorgegeven worden, die bepaalt of de synoniemen als RNA-codons (True) of als DNA-codons (False) moeten teruggegeven worden. Als er niet expliciet een waarde wordt doorgegeven aan de parameter RNA dan is die True als er een U of u voorkomt in codon $$c$$ en False als dat niet het geval is.
-
Schrijf een functie codonoptimalisatie waaraan een collectie (list, tuple of set) van codons (str; DNA of RNA; hoofdletters of kleine letters) moet doorgegeven worden. De functie moet het codon (str) uit de collectie teruggeven waarin het meest C's en G's voorkomen. Als er meerdere codons in de collectie voorkomen die het meest C's en G's bevatten, dan nemen we daarvan het codon dat alfabetisch gezien het eerst komt (zonder onderscheid te maken tussen hoofdletters en kleine letters). We noemen dit het optimaal codon uit de collectie codons.
-
Schrijf een functie peptideoptimalisatie waaraan twee argumenten moeten doorgegeven worden: i) een peptide $$p$$ (str; DNA of RNA; hoofdletters of kleine letters) en ii) de dictionary-voorstelling van een genetische code $$\mathcal{C}$$. De functie moet de geoptimaliseerde versie (str; in hoofdletters) van peptide $$p$$ teruggeven, waarin elk codon $$c$$ vervangen werd door het optimaal codon uit de synoniemen van codon $$c$$ volgens genetische code $$\mathcal{C}$$. Als peptide $$p$$ op het einde één of twee basen heeft die geen deel uitmaken van een codon, dan blijven die ongewijzigd in de geoptimaliseerde versie. De functie heeft ook nog een optionele parameter RNA waaraan een Booleaanse waarde (bool) kan doorgegeven worden, die bepaalt of de geoptimaliseerde versie als RNA-peptide (True) of als DNA-peptide (False) moet teruggegeven worden. Als er niet expliciet een waarde wordt doorgegeven aan de parameter RNA dan is die True als er een U of u voorkomt in peptide $$p$$ en False als dat niet het geval is.
Voorbeeld
In onderstaande voorbeeldsessie gaan we ervan uit dat het tekstbestand genetische_code.txt3 zich in de huidige directory bevindt.
>>> code = genetische_code('genetische_code.txt4')
>>> code['TCT']
'S'
>>> code['TCA']
'S'
>>> code['ATG']
'M'
>>> omgekeerde_code = omgekeerde_genetische_code(code)
>>> omgekeerde_code['S']
{'TCA', 'TCC', 'TCT', 'AGC', 'TCG', 'AGT'}
>>> omgekeerde_code['M']
{'ATG'}
>>> omgekeerde_code['*']
{'TGA', 'TAA', 'TAG'}
>>> synoniemen('TCT', code)
{'TCC', 'AGT', 'TCT', 'AGC', 'TCA', 'TCG'}
>>> synoniemen('atg', code, RNA=True)
{'AUG'}
>>> synoniemen('UGA', code)
{'UAA', 'UAG', 'UGA'}
>>> codonoptimalisatie(['tca', 'tcc', 'tct', 'agc', 'tcg', 'agt'])
'agc'
>>> codonoptimalisatie({'ATG'})
'ATG'
>>> codonoptimalisatie(('UAA', 'UAG', 'UGA'))
'UAG'
>>> peptideoptimalisatie('ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTT', code)
'ATGTTCGTCTTCCTCGTCCTCCTCCCCCTCGTCAGCAGCCAGTGCGTC'
>>> peptideoptimalisatie('AUGUUUGUUUUUCUUGUUUUAUUGCCACUAGUCUCUAGUCAGUGUGUU', code, RNA=True)
'AUGUUCGUCUUCCUCGUCCUCCUCCCCCUCGUCAGCAGCCAGUGCGUC'