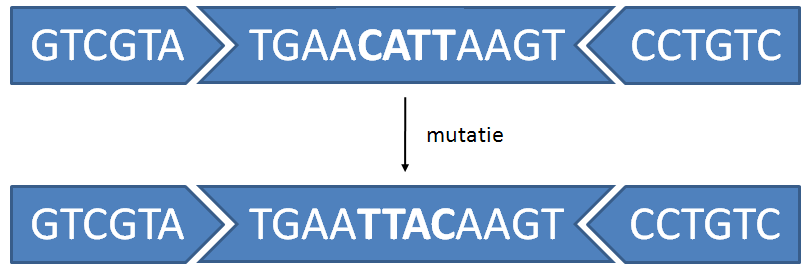
DNA sequenties kunnen voorgesteld worden door strings die enkel bestaan uit de letters A, C, G en T. Een bepaalde vorm van mutaties in het DNA komt voor wanneer een deelsequentie van het DNA volledig wordt omgekeerd tijdens het replicatieproces. Normaal gezien doen dergelijke omkeringen (reversals) zich voor tussen wat men omgekeerde paren (inverted pairs) noemt. Wanneer het patroon TGAA bijvoorbeeld verderop wordt gevolgd door het omgekeerde patroon AAGT, dan is het mogelijk dat het segment van het DNA dat tussen deze patronen zit ingesloten, wordt omgekeerd en terug wordt aangehecht, omdat de bindingen aan beide uiteinden lokaal dezelfde zijn.
Opgave
Schrijf een functie mutatie die voor een gegeven DNA sequentie en een gegeven patroon (beide moeten als argument aan de functie doorgegeven worden) de gemuteerde DNA sequentie teruggeeft. De functie moet hiervoor eerst de meest linkse positie van het patroon binnen de sequentie bepalen, en ook het volgende voorkomen van het omgekeerde patroon. De gemuteerde DNA sequentie wordt dan bekomen door het segment tussen het omgekeerde paar om te keren. De functie moet de originele (niet-gemuteerde) DNA sequentie teruggeven indien het patroon niet voorkomt in de originele DNA sequentie, of indien het niet gevolgd wordt door een omgekeerd patroon.
Voorbeeld
>>> mutatie('GTCGTATGAACATTAAGTCCTGTC', 'TGAA')
'GTCGTATGAATTACAAGTCCTGTC'
>>> mutatie('GTCGTATGAACATTAAGTCCTGTC', 'GTC')
'GTCCTGAATTACAAGTATGCTGTC'
>>> mutatie('GTCGTATGAACATTAAGTCCTGTC', 'TCG')
'GTCGTATGAACATTAAGTCCTGTC'
De verklaring van de resultaten in bovenstaand voorbeeld vind je terug in onderstaand tabel, waarin de omgekeerde paren onderlijnd worden.
| SEQUENTIE | PATROON | RESULTAAT |
|---|---|---|
| GTCGTATGAACATTAAGTCCTGTC | TGAA | GTCGTATGAATTACAAGTCCTGTC |
| GTCGTATGAACATTAAGTCCTGTC | GTC | GTCCTGAATTACAAGTATGCTGTC |
| GTCGTATGAACATTAAGTCCTGTC | TCG | GTCGTATGAACATTAAGTCCTGTC |