Two-sample t-test
Een two-sample t-test is een statistische toets die werd ontwikkeld om verschillen in gemiddelde te detecteren tussen twee onafhankelijke groepen. We introduceren eerst een motiverende dataset. Men vermoedt dat hinderlijke geur onder de oksels (bromhidrosis) wordt veroorzaakt door specifieke microorganismen die behoren tot de groep van de Corynebacterium spp.. Het is immers niet het zweet dat de geur veroorzaakt, maar de geur is het resultaat van specifieke bacteriën die het zweet metaboliseren. Een andere sterk abundante groep wordt gevormd door de Staphylococcus spp.. In de CMET onderzoeksgroep van de Universiteit Gent wordt onderzoek verricht naar de mogelijkheid van microbiële transplanties in de oksels om mensen van de hinderlijke okselgeur te verlossen. Deze therapie bestaat erin om eerst het oksel-microbioom te verwijderen door een lokale antibiotica behandeling, en vervolgens via een microbiële transplantie de populatie te beïnvloeden. (zie: https://youtu.be/9RIFyqLXdVw )
De primaire onderzoeksvraag: leidt de microbiële transplantatie na zes weken tot een verandering in de relatieve abundantie van Staphylococcus spp. in het oksel microbioom in vergelijking met een placebo behandeling die enkel bestaat uit een antibiotica behandeling? Twintig personen met een hinderlijke okselgeur worden willekeurig toegekend aan twee behandelingsgroepen: placebo (enkel antibiotica) en transplantie (antibiotica, gevolgd door microbiële transplantatie). Zes weken na de start van de behandeling wordt een staal van de huid uit de okselholte genomen en worden de relatieve abundanties van Staphylococcus spp. en Corynebacterium spp. in het microbioom gemeten via DGGE (Denaturing Gradient Gel Electrophoresis).
De dataset bevat de variabelen Staph en Cor die de relatieve abundanties (%) weergeven van Staphylococcus spp. en Corynebacterium spp. De variabele Rel werd berekend als
\[\text{Rel}=\frac{\text{Staph}}{\text{Staph}+\text{Cor}}.\]Deze variabele is het relatief aandeel van Staphylococcus spp. op het totaal aantal Staphylococcus spp. en Corynebacterium spp..
We gaan hiervoor opnieuw de microbioom data inlezen. De resultaten worden weergegeven in Figuur 23.
ap <- read_csv("https://raw.githubusercontent.com/GTPB/PSLS20/master/data/armpit.csv")
head(ap)
## # A tibble: 6 x 2
## trt rel
## <chr> <dbl>
## 1 placebo 55.0
## 2 placebo 31.8
## 3 placebo 41.1
## 4 placebo 59.5
## 5 placebo 63.6
## 6 placebo 41.5
ap %>%
ggplot(aes(x = trt, y = rel)) +
geom_boxplot() +
geom_jitter() +
xlab("Relatieve abundantie") +
ylab("Behandeling")
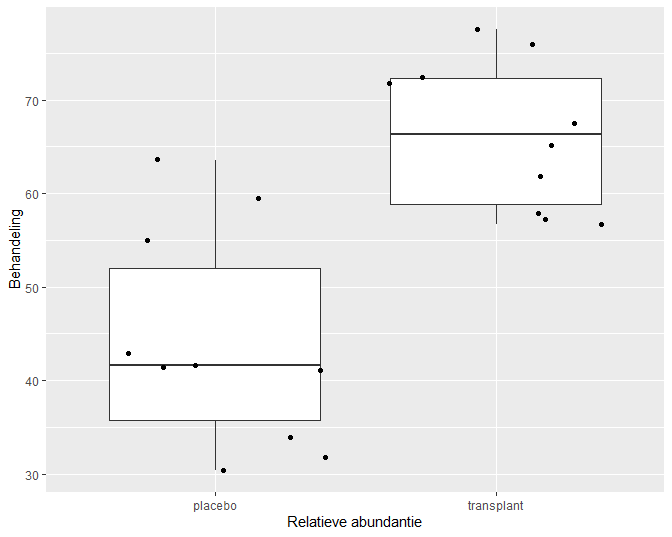
Figuur 23: Boxplot van de relatieve Staphylococcus spp. abundantie t.o.v. het totaal van Staphylococcus spp. en Corynebacterium spp., voor beide behandelingsgroepen.
ap$trt <- as.factor(ap$trt) #zet charactervector om in factor
ap %>%
ggplot(aes(sample = rel)) +
geom_qq() +
geom_qq_line() +
facet_grid(.~trt) +
ylab("Relatieve abundantie")
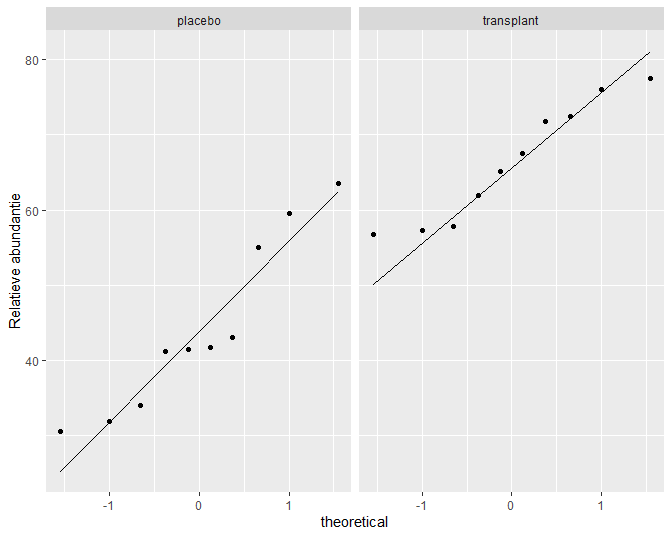
Figuur 24: QQ-plots van relatieve Staphylococcus spp. abundantie t.o.v. het totaal van Staphylococcus spp. en Corynebacterium spp.
Normaliteit van de data in beide groepen wordt ook nagegaan d.m.v. QQ-plots (zie Figuur 24). De QQ-plots geven geen te grote afwijkingen weer van normaliteit.
We introduceren eerst de notatie.