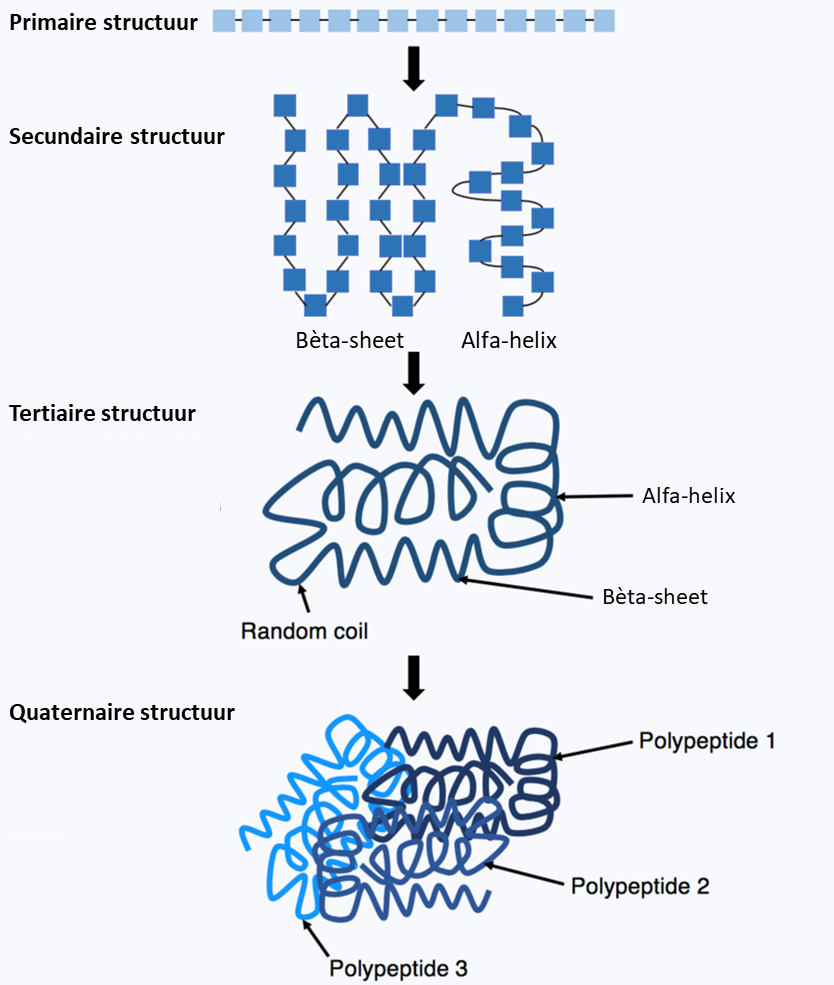
Een eiwit1 bestaat uit een lineaire keten van aminozuren2. In een proces dat eiwitopvouwing3 genoemd wordt, vouwt die keten zich spontaan op tot een natieve4 driedimensionale structuur5 waarin het eiwit biologisch werkzaam is.
DNA-sequenties bevatten informatie om ketens van aminozuren te vormen, maar het opvouwen en structureren van de eiwitten is een fysisch proces dat niet direct uit DNA kan afgeleid worden. Om de structuur te bepalen waarin een eiwit zich opvouwt, kijken wetenschappers voornamelijk naar experimentele technieken zoals röntgenkristallografie6, cryo-elektronenmicroscopie7 en kernspinresonantie8. Dit zijn echter bijzonder dure en tijdrovende technieken.
De afgelopen 60 jaar konden wetenschappers de structuren van ruim 170.000 eiwitten in kaart brengen. Bij de verschillende levensvormen op aarde zijn er echter meer dan 200 miljoen eiwitten bekend. Om die allemaal te kunnen identificeren, wordt koortsachtig gezocht naar computationele methoden waarmee de eiwitstructuur veel sneller kan voorspeld worden.
Tot nu toe kwam hun nauwkeurigheid echter verre van in de buurt van experimentele technieken, waardoor ze nog van weinig praktisch nut geweest zijn. Maar in 2018 werd de wetenschappelijke wereld met verstomming geslagen door de nauwkeurigheid waarmee AlphaFold9 eiwitstructuren kon voorspellen. Dit algoritme werd ontwikkeld door Google DeepMind10 en met behulp van artificiële intelligentie doet het er eerst een paar weken over om uit de 170.000 gekende eiwitstructuren te leren hoe eiwitopvouwing werkt, waarna het telkens slechts een paar dagen nodig heeft om nauwkeurige voorspellingen van onbekende eiwitstructuren te maken.
Opgave
De Protein Data Bank11 (PDB) is een vrij toegankelijke databank met gegevens over de driedimensionale structuur van eiwitten (of andere soorten biomoleculen zoals nucleïnezuren). De driedimensionale structuur wordt beschreven in het zogenaamde PDB-formaat: een formaat voor tekstbestanden (met extensie .pdb) die er bijvoorbeeld als volgt uitzien:
HEADER EXTRACELLULAR MATRIX 22-JAN-98 1A3I TITLE X-RAY CRYSTALLOGRAPHIC DETERMINATION OF A COLLAGEN-LIKE TITLE 2 PEPTIDE WITH THE REPEATING SEQUENCE (PRO-PRO-GLY) … EXPDTA X-RAY DIFFRACTION AUTHOR R.Z.KRAMER,L.VITAGLIANO,J.BELLA,R.BERISIO,L.MAZZARELLA, AUTHOR 2 B.BRODSKY,A.ZAGARI,H.M.BERMAN … REMARK 350 BIOMOLECULE: 1 REMARK 350 APPLY THE FOLLOWING TO CHAINS: A, B, C REMARK 350 BIOMT1 1 1.000000 0.000000 0.000000 0.00000 REMARK 350 BIOMT2 1 0.000000 1.000000 0.000000 0.00000 … SEQRES 1 A 9 PRO PRO GLY PRO PRO GLY PRO PRO GLY SEQRES 1 B 6 PRO PRO GLY PRO PRO GLY SEQRES 1 C 6 PRO PRO GLY PRO PRO GLY … ATOM 1 N PRO A 1 8.316 21.206 21.530 1.00 17.44 N ATOM 2 CA PRO A 1 7.608 20.729 20.336 1.00 17.44 C ATOM 3 C PRO A 1 8.487 20.707 19.092 1.00 17.44 C ATOM 4 O PRO A 1 9.466 21.457 19.005 1.00 17.44 O ATOM 5 CB PRO A 1 6.460 21.723 20.211 1.00 22.26 C … HETATM 130 C ACY 401 3.682 22.541 11.236 1.00 21.19 C HETATM 131 O ACY 401 2.807 23.097 10.553 1.00 21.19 O HETATM 132 OXT ACY 401 4.306 23.101 12.291 1.00 21.19 O …
Elke regel van een PDB-bestand heeft een bepaald type, aangegeven door de eerste zes karakters van de regel (posities 1–6). Wat ons hier interesseert zijn de ATOM-regels (regels met type ATOM) die elk één atoom beschrijven in de driedimensionale structuur van het eiwit. Op posities 31–38, 39–46 en 47–54 staan reële getallen die de driedimensionale coördinaten van het atoom in de eiwitstructuur aangeven (uitgedrukt in ångström12). We zullen dergelijke coördinaten voorstellen als een tuple $$(x, y, z)$$ van drie reële getallen (float). Op posities 77–78 staat de symbolische voorstelling van het atoom (in hoofdletters). Elk informatieveld waarvan we de begin- en eindposities hebben aangegeven, kan vooraan en/of achteraan ook spaties bevatten. Onderstaande tabel geeft nog eens een samenvatting van de informatie uit de ATOM-regels die we voor deze opgave nodig hebben.
| karakters | formaat | beschrijving |
|---|---|---|
| 1–6 | tekst | soort regel (in dit geval de tekst ATOM) |
| 31–38 | reëel getal | $$x$$-coördinaat van het atoom |
| 39–46 | reëel getal | $$y$$-coördinaat van het atoom |
| 47–54 | reëel getal | $$z$$-coördinaat van het atoom |
| 77–78 | tekst | symbolische voorstelling van het atoom (in hoofdletters) |
Voor de berekening van sommige eigenschappen van eiwitstructuren hebben we ook de massa van de atomen nodig. Die is terug te vinden in een tekstbestand met de volgende vorm:
atoomnummer symbool engelse naam nederlandse naam atoommassa 1 H hydrogen waterstof 1.00794 2 He helium helium 4.002602 3 Li lithium lithium 6.941 4 Be beryllium beryllium 9.012182 5 B boron boor 10.811 6 C carbon koolstof 12.011 7 N nitrogen stikstof 14.00674 8 O oxygen zuurstof 15.9994 9 F fluorine fluor 18.9984 10 Ne neon neon 20.1797 …
De eerste regel is een hoofding (en mag dus genegeerd worden). Elke volgende regel beschrijft een atoom aan de hand van vijf informatievelden die van elkaar gescheiden worden door tabs: i) atoomnummer, ii) symbolische voorstelling (correspondeert met het laatste veld van de ATOM-regels uit een PDB-bestand), iii) Engelse naam, iv) Nederlandse naam en v) atoommassa (een reeël getal). Gevraagd wordt:
-
Schrijf een functie afstand waaraan twee driedimensionale coördinaten $$(x_1, y_1, z_1)$$ en $$(x_2, y_2, z_2)$$ moet doorgegeven worden. De functie moet de Euclidische afstand tussen de twee coördinaten teruggeven, die berekend wordt als: \[ \sqrt{(x_1 - x_2)^2 + (y_1 - y_2)^2 + (z_1 - z_2)^2} \]
-
Schrijf een functie lees_atomen waaraan de locatie (str) van een PDB-bestand moet doorgegeven worden. De functie moet een lijst (list) met de atomen uit het PDB-bestand teruggeven, opgelijst in de volgorde waarin ze in het PDB-bestand voorkomen. Elk atoom correspondeert met een ATOM-regel en wordt voorgesteld als een tuple met twee elementen: i) de symbolische voorstelling van het atoom (str; zonder spaties vooraan en achteraan) en ii) de driedimensionele coördinaten van het atoom.
-
Schrijf een functie lees_massa waaraan de locatie (str) van een tekstbestand met de massa van de atomen moet doorgegeven worden. De functie moet een dictionary (dict) teruggeven, die de symbolische voorstelling van elk atoom (str; in hoofdletters) afbeeldt op zijn massa (float).
-
Schrijf een functie eiwitmassa waaraan de locaties (str) van twee tekstbestanden moeten doorgegeven worden: i) een PDB-bestand met de driedimensionale structuur van een eiwit en ii) een bestand met de massa van de atomen. De functie moet de totale massa (float) van het eiwit teruggeven. Voor een eiwit dat bestaat uit $$n$$ atomen met massa $$m_i$$ ($$1 \leq i \leq n$$) wordt de totale massa berekend als \[ \sum_{i=1}^{n}m_i \]
-
Schrijf een functie massacentrum waaraan de locaties (str) van twee tekstbestanden moeten doorgegeven worden: i) een PDB-bestand met de driedimensionale structuur van een eiwit en ii) een bestand met de massa van de atomen. De functie moet het massacentrum van het eiwit teruggeven. Voor een eiwit dat bestaat uit $$n$$ atomen met massa $$m_i$$ ($$1 \leq i \leq n$$) en coördinaten $$(x_i, y_i, z_i)$$ is het massacentrum een coördinaat $$(x, y, z)$$ met \[ \, x = \dfrac{\displaystyle\sum_{i=1}^n m_i\,x_i}{\displaystyle\sum_{i=1}^n m_i} \qquad \, y = \dfrac{\displaystyle\sum_{i=1}^n m_i\,y_i}{\displaystyle\sum_{i=1}^n m_i} \qquad \, z = \dfrac{\displaystyle\sum_{i=1}^n m_i\,z_i}{\displaystyle\sum_{i=1}^n m_i} \]
Voorbeeld
In onderstaande voorbeeldsessie gaan we ervan uit dat de tekstbestanden mbs.pdb13 en periodiek_systeem.txt14 zich in de huidige directory bevinden.
>>> afstand((15.74, 11.178, -11.733), (15.234, 10.462, -10.556))
1.4676583389876559
>>> atomen = lees_atomen('mbs.pdb15')
>>> len(atomen)
1223
>>> atomen[0]
('N', (15.74, 11.178, -11.733))
>>> atomen[1]
('C', (15.234, 10.462, -10.556))
>>> atomen[-1]
('O', (-11.704, -9.2, 0.489))
>>> massa = lees_massa('periodiek_systeem.txt16')
>>> massa['H']
1.00794
>>> massa['O']
15.9994
>>> massa['FE']
55.847
>>> eiwitmassa('mbs.pdb17', 'periodiek_systeem.txt18')
16036.10434000035
>>> massacentrum('mbs.pdb19', 'periodiek_systeem.txt20')
(13.77160318215512, -2.956867920527567, 7.905965346916511)