Het bepalen van het volledige genoom van een organisme (genoomsequenering) is één van de hoekstenen van de bioinformatica. Helaas beschikken we nog steeds niet over microscopische technologie die toelaat om tot op nucleotideniveau in te zoomen en zo de reeks nucleotiden van een genoom één voor één af te lezen. Onderzoekers beschikken echter wel over chemische methoden die kunnen toegepast worden voor het genereren en identificeren van kortere DNA fragmenten. In deze context worden de korte fragmenten reads genoemd. Nadat we een grote collectie van reads geïdentificeerd hebben uit meerdere kopieën van hetzelfde genoom, blijft de uitdaging om het genoom te reconstrueren uit deze korte stukjes DNA. Dit proces wordt genoomassemblage genoemd.
Ondanks het feit dat genoomsequenering vandaag de dag is uitgegroeid tot een miljardenbusiness, blijven de sequeneringsmachines die de reads identificeren nog steeds in een substantieel aantal gevallen fouten produceren. Om het nog erger te maken, zijn deze fouten ook nog eens onvoorspelbaar. Het is moeilijk om te bepalen of de machine een fout gemaakt heeft, laat staan om te bepalen waar de fout voorkomt in de read. Daarom is foutcorrectie van de reads meestal een belangrijke eerste stap bij genoomassemblage. Zoals dat ook met puntmutaties het geval is, komen fouten waarbij één enkele nucleotide van de read verkeerd geïnterpreteerd wordt het vaakst voor.
Opgave
Gegeven is een collectie van reads die allemaal even lang zijn. Voor elke read $$r$$ in deze collectie is één van de volgende uitspraken van toepassing:
-
$$r$$ werd correct gesequeneerd en komt minstens twee keer voor in de dataset (mogelijk als een invers complement1)
-
$$r$$ werd verkeerd gesequeneerd (de read komt slechts één keer voor in de dataset) en er is juist één correcte read (of het invers complement van die read) in de dataset waarvoor de Hammingafstand2 ten opzichte van de verkeerde read gelijk is aan 1; in dit geval kan de correcte read (of zijn invers complement) beschouwd worden als de foutcorrectie van de verkeerde read
-
$$r$$ werd verkeerd gesequeneerd (de read komt slechts één keer voor in de dataset) en heeft Hammingafstand minstens 2 met alle correcte reads en het invers complement van alle correcte reads in de dataset; in dat geval wordt de verkeerde read een wees genoemd waarvoor geen foutcorrectie kan gesuggereerd worden
Je opdracht bestaat erin om de verweesde reads te bepalen en om de foutcorrecties te bepalen voor de andere verkeerde reads. Hiervoor ga je als volgt te werk:
-
Schrijf een functie hamming waaraan twee strings moeten doorgegeven worden. De functie moet een AssertionError opwerpen met de boodschap strings moeten even lang zijn indien de gegeven strings niet dezelfde lengte hebben. Anders moet de functie de Hammingafstand tussen beide strings teruggeven.
-
Schrijf een functie complement waaraan een DNA sequentie moet doorgegeven worden (dit is een string die enkel bestaat uit de hoofdletters A, C, G en T). De functie moet het invers complement van de gegeven DNA sequentie teruggeven.
-
Schrijf een functie normaalvorm waaraan een DNA sequentie moet doorgegeven worden. De functie moet ofwel de gegeven sequentie zelf teruggeven, of diens invers complement, afhankelijk van welke string alfabetisch eerst komt. We noemen die string de normaalvorm van de DNA sequentie.
-
Schrijf een functie voorkomens waaraan een collectie (een lijst, tuple, …) van reads moet doorgegeven worden. De functie moet een dictionary teruggeven die de normaalvorm van elke read uit de collectie afbeeldt op het aantal keer dat een read met die normaalvorm voorkomt in de gegeven collectie.
-
Schrijf een functie fouten waaraan een collectie (een lijst, tuple, …) van reads moet doorgegeven worden. De functie mag hierbij veronderstellen dat alle reads dezelfde lengte hebben. De functie moet een tuple met twee elementen teruggeven. Het eerste element is de verzameling van verweesde reads uit de gegeven collectie. Het tweede element is de alfabetisch gesorteerde lijst van alle andere verkeerde reads. Elke verkeerde read uit die lijst wordt voorgesteld als een tuple dat — naast de verkeerde read zelf — als tweede element ook de correctie van de read bevat. Het sorteren moet enkel gebeuren op basis van de verkeerde reads.
Invers complement
Het invers complement van een DNA sequentie wordt verkregen door de string om te keren en het complement te nemen van elke base (A en T zijn complementaire basen, net zoals C en G). Naast het nemen van het complement moeten we de string ook omkeren omwille van de gerichtheid van DNA. DNA-replicatie en -transcriptie gebeuren immers van het 5' uiteinde tot het 3' uiteinde, en het 3' uiteinde van de ene streng ligt tegenover het 5' uiteinde van de complementaire streng. Als we dus gewoon het complement zouden nemen, dan zou de tweede streng in de verkeerde richting gelezen worden.

Hammingafstand
De Hammingafstand tussen twee strings met dezelfde lengte is het minimaal aantal karakters dat moet vervangen worden om de ene string om te zetten in de andere. Als de strings worden aangeduid door $$s_1$$ en $$s_2$$, dan wordt de Hammingafstand tussen beide strings genoteerd als $$d_H(s_1,s_2)$$.
De berekening van de Hammingafstand kan makkelijk visueel voorgesteld worden: de afstand tussen twee strings is immers niets anders dan het aantal posities waarop verschillende karakters voorkomen in beide strings. Zo is de Hammingafstand tussen de twee strings die hieronder weergegeven worden bijvoorbeeld gelijk aan 7.
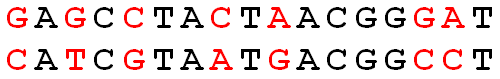
Voorbeeld
>>> hamming('TTCAT', 'TTCAT')
0
>>> hamming('TTCAT', 'TTGAT')
1
>>> hamming('TTCAT', 'GAGGA')
5
>>> hamming('TTCAT', 'TTT')
Traceback (most recent call last):
AssertionError: strings moeten even lang zijn
>>> complement('TTCAT')
'ATGAA'
>>> complement('TTGAT')
'ATCAA'
>>> complement('GAGGA')
'TCCTC'
>>> normaalvorm('TTCAT')
'ATGAA'
>>> normaalvorm('TTGAT')
'ATCAA'
>>> normaalvorm('GAGGA')
'GAGGA'
>>> reads = ['TCATC', 'TTCAT', 'TCATC', 'TGAAA', 'GAGGA', 'TTTCA', 'ATCAA', 'TTGAT', 'AGGCT']
>>> voorkomens(reads)
{'GAGGA': 1, 'ATGAA': 1, 'TGAAA': 2, 'GATGA': 2, 'AGCCT': 1, 'ATCAA': 2}
>>> fouten(reads)
({'AGGCT'}, [('GAGGA', 'GATGA'), ('TTCAT', 'TTGAT')])
>>> reads = ('GATTA', 'GATTA', 'TAATC', 'GAATC', 'GATTA', 'TAAGC', 'TAATA')
>>> voorkomens(reads)
{'GATTA': 4, 'TAATA': 1, 'GCTTA': 1, 'GAATC': 1}
>>> fouten(reads)
(set(), [('GAATC', 'TAATC'), ('TAAGC', 'TAATC'), ('TAATA', 'TAATC')])