Genoomassemblage is het computationele proces waarbij de genoomsequentie van een organisme bepaald wordt. Onderzoekers zijn momenteel nog niet in staat om de nucleotiden van een volledig genoom in één keer te bepalen. In plaats daarvan doen ze een beroep op hoogtechnologische chemische methoden om de basenvolgorde te bepalen van korte stukjes DNA (doorgaans minder dan 1000 bp lang). Deze korte stukjes worden de reads genoemd.
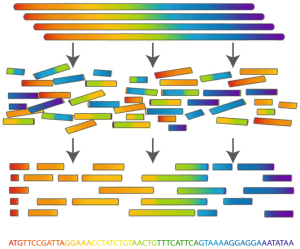
Om een volledig genoom te sequeneren, gaan onderzoekers verschillende kopieën van het genoom als het ware chemisch opblazen, waardoor elk van de bekomen reads door een sequenator kan uitgelezen worden. Om het genoom terug te reconstrueren, wordt gebruikgemaakt van de overlap tussen de reads om te bepalen welke reads naast elkaar op het genoom liggen. Als bijvoorbeeld de korte reads GACCTACA en ACCTACAA uitgelezen worden, dan kunnen we uit het feit dat ze overlappen, afleiden dat beide afkomstig zijn van het DNA fragment GACCTACAA.
In deze opgave lossen we een vereenvoudige versie van genoomassemblage-probleem op, waarbij alle reads vaste lengte $$k$$ hebben ($$k$$-meren), alle $$k$$-meren uit het oorspronkelijke genoom juiste één keer voorkomen en er geen leesfouten voorkomen in de reads.
Opgave
We bepalen eerst alle $$k$$-meren van een woord van lengte $$n$$. Dit zijn de $$n - k + 1$$ overlappende substrings met vaste lengte $$k$$. Daarna schudden we deze $$k$$-meren willekeurig door elkaar, en moet je proberen om het originele woord terug te reconstrueren op basis van de overlap tussen de $$k$$-meren. Beschouw bijvoorbeeld de volgende vier substrings van lengte 3 (hier weergegeven in alfabetische volgorde):
ANA ANA BAN NAN
Dit zijn de vier 3-meren van het woord BANANA. Gevraagd wordt:
-
Schrijf een functie kmeren waaraan twee argumenten moeten doorgegeven worden: i) een string die bestaat uit $$n$$ letters (waarbij $$3 \leq n \leq 100.000$$) $$$$ en ii) een getal $$k \in \mathbb{N}$$ (waarbij $$2 \leq k \leq 15$$ en $$k \leq n$$). De functie moet een lijst teruggeven met de $$n - k + 1$$ overlappende substrings van lengte $$k$$ van de gegeven string. Er is geen opgelegde volgorde waarin deze $$k$$-meren moet opgelijst worden
-
Schrijf een functie assembleer waaraan een lijst van $$k$$-meren moet doorgegeven worden. Hierbij moet elk $$k$$-meer een string zijn die bestaat uit $$k\in \mathbb{N}$$ letters (waarbij $$2 \leq k \leq 15$$). De functie moet een string van lengte $$n \in \mathbb{N}$$ teruggeven waarvan de $$n - k + 1$$ overlappende substrings van lengte $$k$$ corresponderen met de gegeven lijst van $$k$$-meren, los van de volgorde van de $$k$$-meren. Indien het niet mogelijk is om deze string samen te stellen, dan moet een AssertionError opgeworpen worden met de boodschap ongeldige k-meren.
Ondanks het feit dat dit een sterk vereenvoudigde versie is van het echte genoomassemblage-probleem, is het op zich al vrij uitdagend om met een efficiënte oplossing op de proppen te komen. De opdracht bestaat er immers niet enkel in om een implementatie te vinden die minstens één oplossing kan vinden, maar ook om die implementatie zo snel mogelijk te maken. In het artikel van Compeau et al. (2011) vind je de nodige inspiratie om het probleem efficiënt aan te pakken. In de praktijk worden genoomassemblages typisch uitgevoerd op een paar miljoen reads van 40 tot 10.000 baseparen (afhankelijk van de gebruikte technologie voor het sequeneren), waarmee genoomsequenties in grootteorde van miljoenen of miljarden baseparen moeten samengesteld worden.
Voorbeeld
>>> kmeren('banana', 2)
['ba', 'an', 'na', 'an', 'na']
>>> kmeren('cucumber', 2)
['cu', 'uc', 'cu', 'um', 'mb', 'be', 'er']
>>> kmeren('elderberry', 3)
['eld', 'lde', 'der', 'erb', 'rbe', 'ber', 'err', 'rry']
>>> kmeren('pineapple', 3)
['pin', 'ine', 'nea', 'eap', 'app', 'ppl', 'ple']
>>> kmeren('mississippi', 3)
['mis', 'iss', 'ssi', 'sis', 'iss', 'ssi', 'sip', 'ipp', 'ppi']
>>> kmeren('alfalfa', 3)
['alf', 'lfa', 'fal', 'alf', 'lfa']
>>> assembleer(['an', 'an', 'ba', 'na', 'na'])
'banana'
>>> assembleer(['be', 'cu', 'cu', 'er', 'mb', 'uc', 'um'])
'cucumber'
>>> assembleer(['ber', 'der', 'eld', 'erb', 'err', 'lde', 'rbe', 'rry'])
'elderberry'
>>> assembleer(['app', 'eap', 'ine', 'nea', 'pin', 'ple', 'ppl'])
'pineapple'
>>> assembleer(['ipp', 'iss', 'iss', 'mis', 'ppi', 'sip', 'sis', 'ssi', 'ssi'])
'mississippi'
>>> assembleer(['alf', 'alf', 'fal', 'lfa', 'lfa'])
'alfalfa'
Uitdaging
Je hebt nu een algoritme geïmplementeerd waarmee woorden kunnen gereconstrueerd woorden op basis van een willekeurig geordende lijst van al hun $$k$$-meren. De vraag die zich nu stelt is hoe snel en hoe robuust je implementatie is. Het bestand words8.txt bevat een lijst van 70.000+ Engelse woorden van minstens 8 karakters, waarbij elk woord op een afzonderlijke regel staat. Gebruik je implementatie om elk woord in deze lijst uit te lezen, op te splitsen in al zijn $$k$$-meren (kies zelf een vaste $$k$$; hoe kleiner $$k$$ hoe moeilijker het probleem), de $$k$$-meren willekeurig door elkaar te schudden en daarna het oorspronkelijke woord te reconstrueren op basis van de lijst van $$k$$-meren. Analyseer het gedrag van je "assemblage"-programma:
- hoe snel kan je programma alle woorden in het bestand verwerken en reconstrueren?
- hoeveel woorden worden correct gereconstrueerd? hoe definieer je in dit geval "correct?
- in welke omstandigheden faalt het algoritme om het woord te reconstrueren?
- werkt het algoritme nog steeds als enkele $$k$$-meren uit de lijst verwijderd worden? wat als sommige $$k$$-meren meerdere keren gesampled worden? (sampling with replacement)
- kan het algoritme omgaan met fouten die geïntroduceerd worden in sommige $$k$$-meren?
Bronnen
-
Baker M (2012). De novo genome assembly: what every biologist should know. Nature Methods 9, 333-337.
-
Compeau PEC, Philip EC, Pevzner PA, Tesler G (2011). How to apply de Bruijn graphs to genome assembly. Nature Biotechnology 29(11), 987-991.
-
Miller JR, Koren S, Sutton G (2010). Assembly algorithms for next-generation sequencing data. Genomics 95(6), 315-327.
-
Schatz MC, Delcher AL, Salzberg SL (2010). Assembly of large genomes using second-generation sequencing. Genome Research 20, 1165-1173.